M1 BBS Graphes TP Librairies R
From silico.biotoul.fr
m (→visNetwork) |
m (→ggtree) |
||
| Line 13: | Line 13: | ||
</source> | </source> | ||
| - | read.tree au lien de read.newick (ape plus ancienne) | + | |
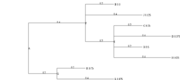
| + | [[Image:Tree_toy_example.png|thumb|Toy example. Télécharger au format [[Media:Tree_toy_example.svg|SVG]] ou [[Media:Tree_toy_example.nw|Newick]].]] | ||
| + | |||
| + | Au format Newick : | ||
| + | ((B:0.2,J:0.4,(C:0.2,D:0.4,H:0.2,I:0.4)E:0.2)F:0.4,(K:0.2,L:0.4)G:0.2)A:0.2; | ||
| + | |||
| + | '''Remarque :''' <tt>read.tree</tt> au lien de <tt>read.newick</tt> (ape plus ancienne) | ||
| + | |||
| + | |||
| + | Premier plot avec les paramètres par défaut | ||
| + | ```{r} | ||
| + | ggtree(t) | ||
| + | ``` | ||
| + | |||
| + | N'affiche que l'arbre, sans les étiquettes ni autre. | ||
| + | |||
| + | <source lang="rsplus"> | ||
| + | ggtree(t)+ geom_tiplab(size=3) | ||
| + | </source> | ||
| + | |||
| + | Et les étiquettes des noeuds internes : | ||
| + | <source lang="rsplus"> | ||
| + | nodelabels = c(t$tip.label, t$node.label) | ||
| + | ggtree(t)+ geom_tiplab(size=3) + geom_text2(aes(subset=!isTip, label=nodelabels), hjust=-.5, size=3) | ||
| + | </source> | ||
| + | |||
| + | Affichage sans prendre en compte les longueurs de branche | ||
| + | <source lang="rsplus"> | ||
| + | ggtree(t, branch.length="none") | ||
| + | </source> | ||
| + | |||
| + | Différents algorithmes de dessin | ||
| + | <source lang="rsplus"> | ||
| + | ggtree(t) + ggtitle("default: rectangular") | ||
| + | ggtree(t, layout="slanted") + ggtitle("slanted") | ||
| + | ggtree(t, layout="circular") + ggtitle("circular") | ||
| + | </source> | ||
| + | |||
| + | Avec ou sans racine | ||
| + | <source lang="rsplus"> | ||
| + | ggtree(t, layout="unrooted") + ggtitle("unrooted layout") | ||
| + | </source> | ||
| + | |||
| + | Avec l'échelle pour les longueurs de branche | ||
| + | <source lang="rsplus"> | ||
| + | ggtree(t) + geom_treescale() | ||
| + | </source> | ||
| + | |||
| + | La même chose avec une thème différent et en stockant le plot dans une variable | ||
| + | <source lang="rsplus"> | ||
| + | p = ggtree(t) + theme_tree2() + ggtitle("rectangular tree with branch lengths and scale") | ||
| + | p | ||
| + | p + geom_tiplab(size=3, color="orange") | ||
| + | </source> | ||
| + | |||
| + | '''Ajout d'annotations''' | ||
| + | |||
| + | Pour cette partie, allez sur https://itol.embl.de/itol.cgi pour télécharger l'arbre au format Newick. | ||
| + | <source lang="rsplus"> | ||
| + | t = read.newick('iTOL.export.newick.txt') | ||
| + | ggtree(t, layout="circular") + ggtitle("iTOL") + geom_tiplab(aes(angle=angle))+ geom_treescale() | ||
| + | </source> | ||
| + | |||
| + | annotations : | ||
| + | <source lang="rsplus"> | ||
| + | t$tip.label[1:10] | ||
| + | t$node.label[1:10] | ||
| + | </source> | ||
| + | |||
| + | Recherche des n° de sommets correspondants aux ancêtres des bactéries, eucaryotes, et archées : | ||
| + | <source lang="rsplus"> | ||
| + | all=c(t$tip.label, t$node.label) | ||
| + | which(all=='Bacteria') | ||
| + | bacteria = which(all=='Bacteria')[1] | ||
| + | which(all=='Eukaryota') | ||
| + | eukaryota = which(all=='Eukaryota')[1] | ||
| + | which(all=='Archaea') | ||
| + | archaea = which(all=='Archaea')[1] | ||
| + | </source> | ||
| + | |||
| + | plot avec l'annotation | ||
| + | <source lang="rsplus"> | ||
| + | ggtree(t, layout="circular") + | ||
| + | ggtitle("iTOL") + | ||
| + | geom_tiplab(aes(angle=angle)) + | ||
| + | geom_hilight(node=bacteria, fill="steelblue", alpha=.1) + | ||
| + | geom_hilight(node=eukaryota, fill="pink", alpha=.1) + | ||
| + | geom_hilight(node=archaea, fill="green", alpha=.1) | ||
| + | </source> | ||
== Graphes == | == Graphes == | ||
Revision as of 13:33, 11 December 2017
Contents |
Arbres
Il existe plusieurs librairies disponibles : ape, genoPlotR, ggtree, ... Celle choisie pour ce TP est ggtree.
ggtree
ggtree est une librairie R/Bioconductor pour la visualisation et l'annotation d'arbres phylogénétiques. Elle est basée sur ggplot2, une librairie pour faire des graphiques plus élaborés qu'avec la fonction plot de R, qu'il vous est conseillé d'expérimenter par vous-même.
Installation de la librairie
source("https://bioconductor.org/biocLite.R") biocLite("ggtree")
Au format Newick :
((B:0.2,J:0.4,(C:0.2,D:0.4,H:0.2,I:0.4)E:0.2)F:0.4,(K:0.2,L:0.4)G:0.2)A:0.2;
Remarque : read.tree au lien de read.newick (ape plus ancienne)
Premier plot avec les paramètres par défaut
```{r}
ggtree(t)
```
N'affiche que l'arbre, sans les étiquettes ni autre.
ggtree(t)+ geom_tiplab(size=3)
Et les étiquettes des noeuds internes :
nodelabels = c(t$tip.label, t$node.label) ggtree(t)+ geom_tiplab(size=3) + geom_text2(aes(subset=!isTip, label=nodelabels), hjust=-.5, size=3)
Affichage sans prendre en compte les longueurs de branche
ggtree(t, branch.length="none")
Différents algorithmes de dessin
ggtree(t) + ggtitle("default: rectangular") ggtree(t, layout="slanted") + ggtitle("slanted") ggtree(t, layout="circular") + ggtitle("circular")
Avec ou sans racine
ggtree(t, layout="unrooted") + ggtitle("unrooted layout")
Avec l'échelle pour les longueurs de branche
ggtree(t) + geom_treescale()
La même chose avec une thème différent et en stockant le plot dans une variable
p = ggtree(t) + theme_tree2() + ggtitle("rectangular tree with branch lengths and scale") p p + geom_tiplab(size=3, color="orange")
Ajout d'annotations
Pour cette partie, allez sur https://itol.embl.de/itol.cgi pour télécharger l'arbre au format Newick.
t = read.newick('iTOL.export.newick.txt') ggtree(t, layout="circular") + ggtitle("iTOL") + geom_tiplab(aes(angle=angle))+ geom_treescale()
annotations :
t$tip.label[1:10] t$node.label[1:10]
Recherche des n° de sommets correspondants aux ancêtres des bactéries, eucaryotes, et archées :
all=c(t$tip.label, t$node.label) which(all=='Bacteria') bacteria = which(all=='Bacteria')[1] which(all=='Eukaryota') eukaryota = which(all=='Eukaryota')[1] which(all=='Archaea') archaea = which(all=='Archaea')[1]
plot avec l'annotation
ggtree(t, layout="circular") + ggtitle("iTOL") + geom_tiplab(aes(angle=angle)) + geom_hilight(node=bacteria, fill="steelblue", alpha=.1) + geom_hilight(node=eukaryota, fill="pink", alpha=.1) + geom_hilight(node=archaea, fill="green", alpha=.1)