M1 MABS BBS Math TD Proba
From silico.biotoul.fr
m (→Vraissemblance) |
m (→Vraissemblance) |
||
| Line 14: | Line 14: | ||
| - | L'[[Media:Maths Proba Alignement RBS. | + | <big><u>Calcul de <math>P(sequence/RBS)</math></u></big> |
| + | |||
| + | L'[[Media:Maths Proba Alignement RBS.fasta|alignement]] des séquences de RBS de ''B. subtilis'' vous est fournit au format FASTA. Il va vous servir à calculer P(sequence/RBS). Pour cela, vous devrez construire la matrice de fréquence de chaque résidu à chaque position (la fréquence observée sert à estimer la probabilité). | ||
| + | |||
| + | Pour lire le fichier FASTA, utilisez la librairie <tt>seqinr</tt> qui est normalement installée : | ||
| + | <source lang='rsplus'> | ||
| + | library(seqinr) | ||
| + | known_rbs = read.fasta('Maths_Proba_Alignement_RBS.fasta') | ||
| + | |||
| + | # affichage | ||
| + | known_rbs | ||
| + | |||
| + | # premiere sequence (objet sequence) | ||
| + | known_rbs[1] | ||
| + | |||
| + | # premiere sequence (sequence en nucleotides) | ||
| + | attr(known_rbs[1], 'name') # identifiant | ||
| + | getSequence(known_rbs[1]) # objet liste | ||
| + | getSequence(known_rbs[1])[[1]] # 1ere sequence sous forme vecteur de caracteres | ||
| + | </source> | ||
| + | |||
| + | Manipulez l'objet <tt>known_rbs</tt> afin d'avoir une matrice <tt>alignment</tt> comme suit : | ||
| + | <source lang='rsplus'> | ||
| + | > head(alignment) | ||
| + | [,1] [,2] [,3] [,4] [,5] | ||
| + | [1,] "g" "g" "a" "g" "g" | ||
| + | [2,] "g" "g" "a" "g" "g" | ||
| + | [3,] "a" "g" "a" "g" "g" | ||
| + | [4,] "g" "g" "g" "t" "g" | ||
| + | [5,] "g" "a" "g" "g" "t" | ||
| + | [6,] "a" "g" "g" "t" "g" | ||
| + | </source> | ||
| + | |||
| + | A partir de cette matrice, calculer la matrice <tt>f_res_pos</tt> qui donne la fréquence d'un résidu à une position. Vous devriez obtenir : | ||
| + | <source lang='rsplus'> | ||
| + | > round(f_res_pos,2) | ||
| + | [,1] [,2] [,3] [,4] [,5] | ||
| + | a 0.34 0.11 0.39 0.42 0.06 | ||
| + | c 0.00 0.00 0.00 0.00 0.00 | ||
| + | g 0.65 0.89 0.60 0.55 0.88 | ||
| + | t 0.01 0.00 0.01 0.03 0.06 | ||
| + | </source> | ||
| + | |||
| + | Vous pouvez à présent définir une fonction qui calcule P(sequence/RBS) : la probabilité d'une séquence de 5 nucléotides sachant qu'elle correspond à un RBS (produit des probabilités des résidus observés à chaque position). | ||
| + | |||
| + | Rappel: pour définir une fonction : | ||
| + | <source lang='rsplus'> | ||
| + | |||
| + | p_seq_rbs = function(s, f = f_res_pos) { # s: sequence à utiliser, f: frequence des résidus à chaque position | ||
| + | # TO DO | ||
| + | } | ||
| + | # test de la fonction: probabilité d'observer la sequence GGAGG sachant que c'est un RBS | ||
| + | p_seq_rbs(c('g','g','a','g','g')) | ||
| + | </source> | ||
| + | |||
| + | |||
| + | <big><u>Calcul de <math>P(RBS)</math></u></big> | ||
Pour la probabilité de présence d'un RBS, il est possible de l'approximer par le nombre de gènes (4,177) ''B. subtilis'' divisé par la taille de son génome (4,215,606 bp). | Pour la probabilité de présence d'un RBS, il est possible de l'approximer par le nombre de gènes (4,177) ''B. subtilis'' divisé par la taille de son génome (4,215,606 bp). | ||
| + | |||
| + | <big><u>Calcul de <math>P(sequence)</math></u></big> | ||
Pour la probabilité d'observer une séquence, on pourra utiliser le produit des fréquences de chaque nucléotide dans le génome [[Media:BsubA.fas.gz]] | Pour la probabilité d'observer une séquence, on pourra utiliser le produit des fréquences de chaque nucléotide dans le génome [[Media:BsubA.fas.gz]] | ||
Revision as of 14:01, 17 October 2014
Vraissemblance
Rappel :
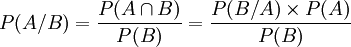
Il s'agit dans la suite de détecter la présence de RBS (Ribosome Binding Site) sur une séquence génomique :
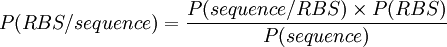
Pour se convaincre de la présence d'un motif conservé, les séquences en amont du site d’initiation de la traduction d'une centaine de séquences de Bacillus subtilis sont fournies (Séquences au format fasta). Elles sont alignées sur le codon start.
A l'aide du site WebLogo, établir le Weblogo correspondant aux séquences de B. subtilis. Où se situent les RBS ?
Calcul de P(sequence / RBS)
L'alignement des séquences de RBS de B. subtilis vous est fournit au format FASTA. Il va vous servir à calculer P(sequence/RBS). Pour cela, vous devrez construire la matrice de fréquence de chaque résidu à chaque position (la fréquence observée sert à estimer la probabilité).
Pour lire le fichier FASTA, utilisez la librairie seqinr qui est normalement installée :
library(seqinr) known_rbs = read.fasta('Maths_Proba_Alignement_RBS.fasta') # affichage known_rbs # premiere sequence (objet sequence) known_rbs[1] # premiere sequence (sequence en nucleotides) attr(known_rbs[1], 'name') # identifiant getSequence(known_rbs[1]) # objet liste getSequence(known_rbs[1])[[1]] # 1ere sequence sous forme vecteur de caracteres
Manipulez l'objet known_rbs afin d'avoir une matrice alignment comme suit :
> head(alignment) [,1] [,2] [,3] [,4] [,5] [1,] "g" "g" "a" "g" "g" [2,] "g" "g" "a" "g" "g" [3,] "a" "g" "a" "g" "g" [4,] "g" "g" "g" "t" "g" [5,] "g" "a" "g" "g" "t" [6,] "a" "g" "g" "t" "g"
A partir de cette matrice, calculer la matrice f_res_pos qui donne la fréquence d'un résidu à une position. Vous devriez obtenir :
> round(f_res_pos,2) [,1] [,2] [,3] [,4] [,5] a 0.34 0.11 0.39 0.42 0.06 c 0.00 0.00 0.00 0.00 0.00 g 0.65 0.89 0.60 0.55 0.88 t 0.01 0.00 0.01 0.03 0.06
Vous pouvez à présent définir une fonction qui calcule P(sequence/RBS) : la probabilité d'une séquence de 5 nucléotides sachant qu'elle correspond à un RBS (produit des probabilités des résidus observés à chaque position).
Rappel: pour définir une fonction :
p_seq_rbs = function(s, f = f_res_pos) { # s: sequence à utiliser, f: frequence des résidus à chaque position # TO DO } # test de la fonction: probabilité d'observer la sequence GGAGG sachant que c'est un RBS p_seq_rbs(c('g','g','a','g','g'))
Calcul de P(RBS)
Pour la probabilité de présence d'un RBS, il est possible de l'approximer par le nombre de gènes (4,177) B. subtilis divisé par la taille de son génome (4,215,606 bp).
Calcul de P(sequence)
Pour la probabilité d'observer une séquence, on pourra utiliser le produit des fréquences de chaque nucléotide dans le génome Media:BsubA.fas.gz
Séquence test
Loi hypergéométrique
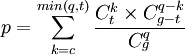
appliquée à la sur-représentation d'une annotation dans un ensemble de gènes, c'est-à-dire à la comparaison de 2 ensembles :
- c: nombre de gènes communs
- q: nombre de gènes du premier ensemble (query par exemple gènes différentiellement exprimés ou co-exprimés)
- t: nombre de gènes du deuxième ensemble (target par exemple gènes annotatés 'biosynthèse des acides aminés')
- g: nombre de gènes dans le génome
A quoi correspondent 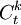 ?
? 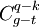 et
et 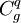 ?
?
Rappel : Combinaisons:
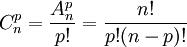
- Calculer la p-valeur pour c=30, q=100, t=300 et g=20000
- Quelle est le plus grand nombre x pour lequel vous pouvez calculer x! ?



