M1 Traitement de Donnees Biologiques - TP 3 R
From silico.biotoul.fr
| Line 30: | Line 30: | ||
</source> | </source> | ||
| - | Question: les proportions observées sont-elles conformes aux proportions mendéliennes? Pour cela il faut répondre aux questions | + | Question: les proportions observées sont-elles conformes aux proportions mendéliennes? Pour cela il faut effectuer la commande suivante, et répondre aux questions ci-après: |
| - | * Quelle est l'hypothèse nulle (H0)du test ? L'hypothèse alternative (H1) ? | + | <source lang='rsplus'> |
| + | chisq.test(freq_obs,p=freq_exp) | ||
| + | </source> | ||
| + | |||
| + | * Quelle est l'hypothèse nulle (H0) du test ? L'hypothèse alternative (H1) ? | ||
* Qu'indique la p-valeur ? | * Qu'indique la p-valeur ? | ||
* La p-valeur est-elle inférieure ou supérieure au seuil alpha = 5% ? | * La p-valeur est-elle inférieure ou supérieure au seuil alpha = 5% ? | ||
| Line 39: | Line 43: | ||
<span style='color: #990000;'>Ajoutez ces parties à votre compte rendu.</span> | <span style='color: #990000;'>Ajoutez ces parties à votre compte rendu.</span> | ||
| + | == Tests de Chi2 de conformité == | ||
| + | <span style='color: #990000;'> Les tests suivant s'intègrent dans le même exercice. </span> | ||
| + | = Test d'adéquation à la loi normale = | ||
| - | |||
| - | + | = Tests d'homogénéité des variances et des moyennes, = | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
Revision as of 14:53, 19 September 2016
Contents |
Tests statistiques
La plupart des tests statistiques courants (et bien d’autres) sont programmés dans R. Dans ce TP, nous allons aborder quelques types de tests statistiques très courants:
- Les tests de Chi2 (test de conformité, test d'indépendance), basés sur les effectifs de différentes classes
- Les tests d'adéquation (ici nous verrons l'adéquation à la loi Normale)
- Les tests d'homogénéité (comparaison de moyennes, comparaison de variances)
ATTENTION: pour chaque test statistique, il est crucial de bien connaître l'hypothèse nulle du test (H0) pour pouvoir correctement interpréter !
Créez tout d'abord un répertoire de travail sur le bureau (par exemple TDB-TP3) et commencez par télécharger le fichier source que vous allez utiliser et compléter pour générer le compte rendu de TP : M1.TDB.TP_tests_R.Rmd (click droit de la souris -- enregistrer la cible sous...). Ouvrez le logiciel RStudio et chargez ce fichier puis lancez sa compilation pour voir le compte rendu. Pour cela cliquez sur le bouton Knit HTML ou bien utilisez la combinaison de touches Ctrl + shift + K.
Tests de Chi2
Tests de Chi2 de conformité
On cherche à tester la conformité d'une (ou plusieurs) valeur(s) par rapport à une (des) valeur(s) standard(s) (ou théoriques). Le test de Chi2 de conformité s'effectue sur les effectifs des différentes classes d'une variable qualitative (ou quantitative).
Notre exemple porte sur les proportion mendéliennes dans une descendance issue d'autofécondation d'une F1 hétérozygote.
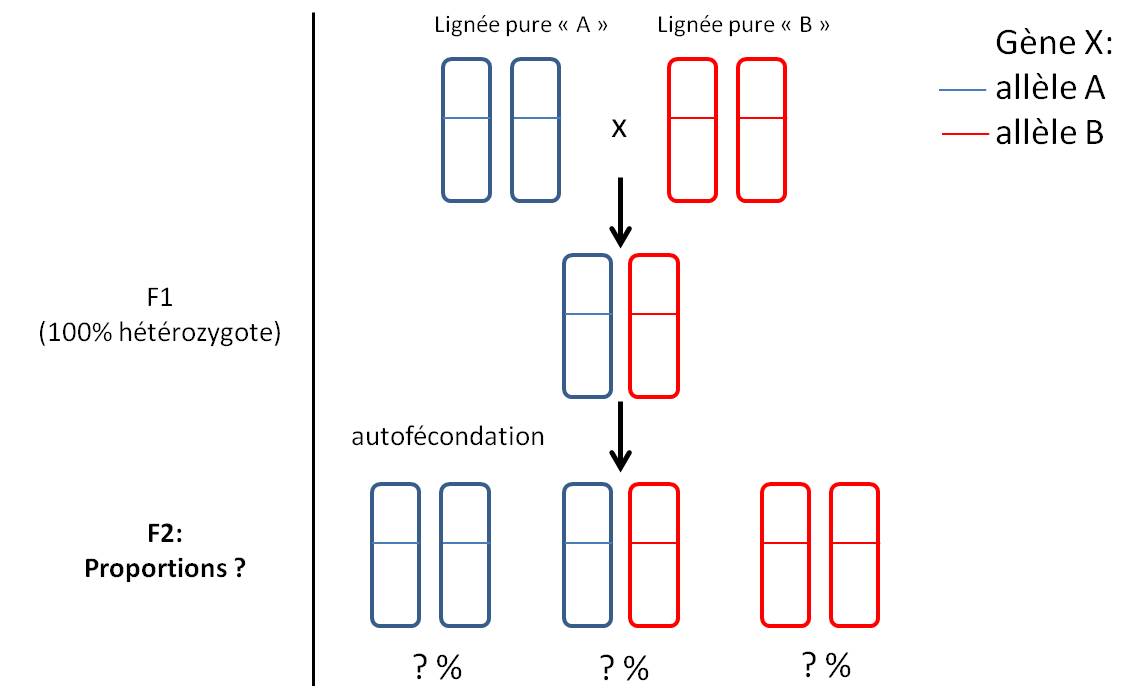
Pour un gène avec 2 allèles, les proportions génotypiques attendues (relatives) dans la descendance sont:
freq_exp =c(?,?,?) # mettre sous forme de fréquences relatives
Suite à un croisement effectué en laboratoire, les proportions génotypiques observées (absolues) parmis 100 individus descendants sont:
freq_obs =c(22,53,25)
Question: les proportions observées sont-elles conformes aux proportions mendéliennes? Pour cela il faut effectuer la commande suivante, et répondre aux questions ci-après:
chisq.test(freq_obs,p=freq_exp)
- Quelle est l'hypothèse nulle (H0) du test ? L'hypothèse alternative (H1) ?
- Qu'indique la p-valeur ?
- La p-valeur est-elle inférieure ou supérieure au seuil alpha = 5% ?
Conclusion:
Ajoutez ces parties à votre compte rendu.
Tests de Chi2 de conformité
Les tests suivant s'intègrent dans le même exercice.
Test d'adéquation à la loi normale
Tests d'homogénéité des variances et des moyennes,
Ajoutez tout cela au compte rendu de TP avant de l'envoyer à votre enseignant par mail (bonhomme@lrsv.ups-tlse.fr ou barriot@biotoul.fr). Le compte rendu est à envoyer avant de commencer le TP4. Envoyez les 2 fichiers (.Rmd et .html). Envoyez-vous aussi le mail en copie pour pouvoir vérifier que tout est bien passé. Mettez un titre tel que "Compte rendu TP3 TDB de -et votre Nom et Prénom-".
Liens
- Site de R : http://www.r-project.org et sites miroirs (dont ceux en France) pour télécharger le logiciel et les librairies : https://cran.r-project.org/mirrors.html
- RStudio : https://www.rstudio.com



