M1 MABS BBS BGPG TD GRNs - 2015
From silico.biotoul.fr
m |
m (moved M1 MABS BBS BGPG TD GRNs to M1 MABS BBS BGPG TD GRNs - 2015) |
||
| (6 intermediate revisions not shown) | |||
| Line 33: | Line 33: | ||
= Principe = | = Principe = | ||
| - | * Calcul d'une matrice d'information mutuelle (ou de corrélations) entre chaque paire de profils d'expression | + | * Calcul d'une matrice d'information mutuelle (ou de corrélations) entre chaque paire de profils d'expression (donc entre chaque paire de gènes) |
** pour cela plusieurs estimateurs sont disponibles et utilisent, pour la plupart, des données nominales. Il faudra donc effectuer une discrétisation au préalable (fonctions fournies dans la librairie). | ** pour cela plusieurs estimateurs sont disponibles et utilisent, pour la plupart, des données nominales. Il faudra donc effectuer une discrétisation au préalable (fonctions fournies dans la librairie). | ||
* Inférence du réseau | * Inférence du réseau | ||
| Line 42: | Line 42: | ||
* Partitionnement du réseau et recherche de fonctions biologiques sur-représentées au sein des clusters | * Partitionnement du réseau et recherche de fonctions biologiques sur-représentées au sein des clusters | ||
| - | = | + | = Constitution du jeu de données = |
* Pour l'inférence : Compendium de données d'expression | * Pour l'inférence : Compendium de données d'expression | ||
| - | L'idée est de récupérer des données d'expression depuis GEO sur l'organisme qui nous intéresse. Ici, on prendra ''Escherichia coli''. Un certain nombre de design de microarray (GPLxxx) sont disponibles. Ceux arborant le plus d'hybridations (GSMxxx) sont [http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL199 GPL199] (52 séries GSExxx et 938 hybridations GSMxxx au 6 février 2013) et [http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc= | + | L'idée est de récupérer des données d'expression depuis GEO sur l'organisme qui nous intéresse. Ici, on prendra ''Escherichia coli''. Un certain nombre de design de microarray (GPLxxx) sont disponibles. Ceux arborant le plus d'hybridations (GSMxxx) sont [http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL199 GPL199] (52 séries GSExxx et 938 hybridations GSMxxx au 6 février 2013) et [http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL3154 GPL3154] (121 séries et 956 hybridations au 6 février 2013 également). |
| - | Après avoir choisi d'utiliser GPL3154, les informations sur les 121 séries ont été étudiées pour sélectionner les plus pertinentes (en fonction de la souche dont proviennent les ARNm extraits, des conditions expérimentales, etc.). Au total, cela fait un peu plus de 800 hybridations. Le fichier GPL et les fichiers GSM ont été téléchargés. Les données ont ensuite été normalisées de manière à pouvoir comparer le niveau d'expression d'un gène dans une condition par rapport à son niveau d'expression dans une autre condition (cf. [[M1_MABS_TDB_TD_Transcriptome_-_Clustering|ici]] pour la procédure de chargement et de normalisation des données au format .CEL d'Affymetrix). Après normalisation, les données ont été prétraités afin d'avoir une matrice gènes-conditions. Pour d'associer un identifiant de spot/probeset à un gène, le fichier donnant les séquences des oligonucléotides des spots a été téléchargé depuis le site d'Affymetrix (le fabricant du microarray). Pour chaque spot, donc à partir des séquences d'oligonucléotides lui correspondant, un blast nucléique a été réalisé sur le génome afin d'identifier quelles étaient les régions complémentaires. Dans le cas où tous les oligonucléotides d'un spot ne pouvaient s'hybrider qu'avec un seul gène, l'association a été effectuée. Le résultat vous est fourni dans le fichier suivant (click droit puis enregistrer sous sinon vous allez afficher le contenu qui fait | + | Après avoir choisi d'utiliser GPL3154, les informations sur les 121 séries ont été étudiées pour sélectionner les plus pertinentes (en fonction de la souche dont proviennent les ARNm extraits, des conditions expérimentales, etc.). Au total, cela fait un peu plus de 800 hybridations. Le fichier GPL et les fichiers GSM ont été téléchargés. Les données ont ensuite été normalisées de manière à pouvoir comparer le niveau d'expression d'un gène dans une condition par rapport à son niveau d'expression dans une autre condition (cf. [[M1_MABS_TDB_TD_Transcriptome_-_Clustering|ici]] pour la procédure de chargement et de normalisation des données au format .CEL d'Affymetrix). Après normalisation, les données ont été prétraités afin d'avoir une matrice gènes-conditions. Pour d'associer un identifiant de spot/probeset à un gène, le fichier donnant les séquences des oligonucléotides des spots a été téléchargé depuis le site d'Affymetrix (le fabricant du microarray). Pour chaque spot, donc à partir des séquences d'oligonucléotides lui correspondant, un blast nucléique a été réalisé sur le génome afin d'identifier quelles étaient les régions complémentaires. Dans le cas où tous les oligonucléotides d'un spot ne pouvaient s'hybrider qu'avec un seul gène, l'association a été effectuée. Le résultat ne contenant que les gènes référencés par RegulonDb vous est fourni dans le fichier suivant (click droit puis enregistrer sous sinon vous allez afficher le contenu qui fait 20Mo): |
| - | [[silico:enseignement/m1-mabs/BGPG/GRNs/ | + | [[silico:enseignement/m1-mabs/BGPG/GRNs/Ecoli.GPL3154.rma.regulondb.txt|Ecoli.GPL3154.rma.regulondb.txt]] |
* Pour la validation : Réseau de référence | * Pour la validation : Réseau de référence | ||
| - | Pour les données de référence, nous allons utiliser celles fournies par [http://regulondb.ccg.unam.mx RegulonDb]. Il s'agit donc de récupérer le réseau mis à disposition (celui entre les facteurs de transcriptions et les | + | Pour les données de référence, nous allons utiliser celles fournies par [http://regulondb.ccg.unam.mx RegulonDb]. Il s'agit donc de récupérer le réseau mis à disposition (celui entre les facteurs de transcriptions et les gènes, c'est-à-dire celui dans la partie Regulatory Network Interactions - TF - gene interactions). Pour simplifier la suite, enregistrez-le sous le nom <tt>network_tf_operon.unfiltered.txt</tt> |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
= Visualisation du jeu de données de référence = | = Visualisation du jeu de données de référence = | ||
Afin de charger le réseau de RegulonDb, il faut convertir le fichier fourni dans un format compréhensible par Cytoscape. Nous utiliserons le format [http://cytoscape.org/manual/Cytoscape2_8Manual.html#SIF%20Format SIF]. | Afin de charger le réseau de RegulonDb, il faut convertir le fichier fourni dans un format compréhensible par Cytoscape. Nous utiliserons le format [http://cytoscape.org/manual/Cytoscape2_8Manual.html#SIF%20Format SIF]. | ||
| - | + | Ecrivez un script Perl pour convertir le fichier fourni par RegulonDb au format SIF. | |
| - | + | ||
| - | + | 'Remarque :' mettre le identifiants en majuscule permettra de ne pas dissocier le gène ( | |
La sortie du script devrait ressembler à cela : | La sortie du script devrait ressembler à cela : | ||
| - | lancement du script: ./regulondb_network-- | + | lancement du script: ./regulondb_network--to--sif.pl network_tf_operon.unfiltered.txt > Ecoli.grn.unfiltered.sif |
| - | début du fichier généré: head | + | début du fichier généré: head Ecoli.grn.unfiltered.sif |
| - | + | ACCB repression ACCB | |
| - | + | ACCB repression ACCC | |
| - | + | ACRR repression ACRA | |
| - | + | ACRR repression ACRB | |
| - | + | ACRR repression ACRR | |
| - | + | ACRR repression MICF | |
| - | + | ADA induction ADA | |
| - | + | ADA repression ADA | |
| - | + | ADA induction AIDB | |
| - | + | ADA induction ALKA | |
| - | + | ||
Chargez le réseau obtenu dans Cytoscape et modifier le style d'affichage pour obtenir quelque chose comme ça : | Chargez le réseau obtenu dans Cytoscape et modifier le style d'affichage pour obtenir quelque chose comme ça : | ||
[[Image:GRNs_Cytoscape_Style.png|link=]] | [[Image:GRNs_Cytoscape_Style.png|link=]] | ||
| - | + | Du fait de l'absence de profil d'expression pour certains gènes, il est nécessaire de filtrer le fichier fourni par RegulonDb pour ne conserver que les gènes ayant un profil (dans le fichier Ecoli.GPL3154.rma.regulondb.txt). Il faut aussi supprimer les boucles (un gène agissant sur lui-même) car celles-ci ne peuvent pas être détectées par les méthodes de reconstruction de réseaux de régulation que nous allons utiliser. Ecrivez un autre script réalisant cette tâche : | |
| - | + | * mémorisation des identifiants ayant un profil, | |
| + | * affichage uniquement des relations impliquant ceux-là | ||
| + | ** sous réserve que le facteur de transcription et la cible soient différents (exemple :ACRR repression ACRR) ET | ||
| + | ** sous réserve que l'on n'ait pas des relations contradictoires (exemple : ADA induction ADA vs. ADA repression ADA). On ne devrait avoir au final que des régulations de type +/induction, -/repression, +-/complex, ?/unknown | ||
| - | + | Script pour le filtrage : | |
| - | + | [[silico:enseignement/m1-mabs/BGPG/GRNs/filter_interactions.pl|filter_interactions.pl]] | |
| + | |||
| + | Utilisation : <tt>perl filter_interactions.pl Ecoli.GPL3154.rma.regulondb.txt network_tf_gene.unfiltered.txt > network_tf_gene.filtered.txt</tt> | ||
= Inférence du réseau de régulation = | = Inférence du réseau de régulation = | ||
| + | |||
Nous allons utiliser maintenant la librairie minet ([http://www.ncbi.nlm.nih.gov/pubmed/18959772 Meyer PE, Lafitte F, Bontempi G., 2008]) pour reconstruire le réseau. Une version plus complète de la vignette (nom de la documentation/tutorial en R) se trouve [[Media:minet.vignette.pdf|ici]]. | Nous allons utiliser maintenant la librairie minet ([http://www.ncbi.nlm.nih.gov/pubmed/18959772 Meyer PE, Lafitte F, Bontempi G., 2008]) pour reconstruire le réseau. Une version plus complète de la vignette (nom de la documentation/tutorial en R) se trouve [[Media:minet.vignette.pdf|ici]]. | ||
Current revision as of 08:28, 25 March 2016
Au cours de ce TD, nous allons utiliser une librairie (minet) faisant partie de la suite Bioconductor afin d'inférer un réseau de régulation à partir de données d'expression.
Une fois le réseau reconstruit, il s'agira d'évaluer sa qualité.
Ensuite, on pourra également visualiser le réseau obtenu avec Cytoscape par exemple.
Contents |
Principe
- Calcul d'une matrice d'information mutuelle (ou de corrélations) entre chaque paire de profils d'expression (donc entre chaque paire de gènes)
- pour cela plusieurs estimateurs sont disponibles et utilisent, pour la plupart, des données nominales. Il faudra donc effectuer une discrétisation au préalable (fonctions fournies dans la librairie).
- Inférence du réseau
- méthodes : CLR, ARACNe, et MRNET
- Evaluation du réseau obtenu par rapport à un jeu de référence
- courbes Precision-Recall et ROC
- Choix d'un seuil, extraction du graphe obtenu et visualisation sous Cytoscape
- Partitionnement du réseau et recherche de fonctions biologiques sur-représentées au sein des clusters
Constitution du jeu de données
- Pour l'inférence : Compendium de données d'expression
L'idée est de récupérer des données d'expression depuis GEO sur l'organisme qui nous intéresse. Ici, on prendra Escherichia coli. Un certain nombre de design de microarray (GPLxxx) sont disponibles. Ceux arborant le plus d'hybridations (GSMxxx) sont GPL199 (52 séries GSExxx et 938 hybridations GSMxxx au 6 février 2013) et GPL3154 (121 séries et 956 hybridations au 6 février 2013 également).
Après avoir choisi d'utiliser GPL3154, les informations sur les 121 séries ont été étudiées pour sélectionner les plus pertinentes (en fonction de la souche dont proviennent les ARNm extraits, des conditions expérimentales, etc.). Au total, cela fait un peu plus de 800 hybridations. Le fichier GPL et les fichiers GSM ont été téléchargés. Les données ont ensuite été normalisées de manière à pouvoir comparer le niveau d'expression d'un gène dans une condition par rapport à son niveau d'expression dans une autre condition (cf. ici pour la procédure de chargement et de normalisation des données au format .CEL d'Affymetrix). Après normalisation, les données ont été prétraités afin d'avoir une matrice gènes-conditions. Pour d'associer un identifiant de spot/probeset à un gène, le fichier donnant les séquences des oligonucléotides des spots a été téléchargé depuis le site d'Affymetrix (le fabricant du microarray). Pour chaque spot, donc à partir des séquences d'oligonucléotides lui correspondant, un blast nucléique a été réalisé sur le génome afin d'identifier quelles étaient les régions complémentaires. Dans le cas où tous les oligonucléotides d'un spot ne pouvaient s'hybrider qu'avec un seul gène, l'association a été effectuée. Le résultat ne contenant que les gènes référencés par RegulonDb vous est fourni dans le fichier suivant (click droit puis enregistrer sous sinon vous allez afficher le contenu qui fait 20Mo):
Ecoli.GPL3154.rma.regulondb.txt
- Pour la validation : Réseau de référence
Pour les données de référence, nous allons utiliser celles fournies par RegulonDb. Il s'agit donc de récupérer le réseau mis à disposition (celui entre les facteurs de transcriptions et les gènes, c'est-à-dire celui dans la partie Regulatory Network Interactions - TF - gene interactions). Pour simplifier la suite, enregistrez-le sous le nom network_tf_operon.unfiltered.txt
Visualisation du jeu de données de référence
Afin de charger le réseau de RegulonDb, il faut convertir le fichier fourni dans un format compréhensible par Cytoscape. Nous utiliserons le format SIF.
Ecrivez un script Perl pour convertir le fichier fourni par RegulonDb au format SIF.
'Remarque :' mettre le identifiants en majuscule permettra de ne pas dissocier le gène (
La sortie du script devrait ressembler à cela :
lancement du script: ./regulondb_network--to--sif.pl network_tf_operon.unfiltered.txt > Ecoli.grn.unfiltered.sif début du fichier généré: head Ecoli.grn.unfiltered.sif ACCB repression ACCB ACCB repression ACCC ACRR repression ACRA ACRR repression ACRB ACRR repression ACRR ACRR repression MICF ADA induction ADA ADA repression ADA ADA induction AIDB ADA induction ALKA
Chargez le réseau obtenu dans Cytoscape et modifier le style d'affichage pour obtenir quelque chose comme ça :
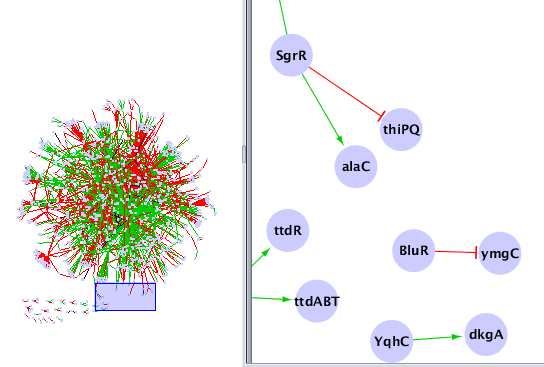
Du fait de l'absence de profil d'expression pour certains gènes, il est nécessaire de filtrer le fichier fourni par RegulonDb pour ne conserver que les gènes ayant un profil (dans le fichier Ecoli.GPL3154.rma.regulondb.txt). Il faut aussi supprimer les boucles (un gène agissant sur lui-même) car celles-ci ne peuvent pas être détectées par les méthodes de reconstruction de réseaux de régulation que nous allons utiliser. Ecrivez un autre script réalisant cette tâche :
- mémorisation des identifiants ayant un profil,
- affichage uniquement des relations impliquant ceux-là
- sous réserve que le facteur de transcription et la cible soient différents (exemple :ACRR repression ACRR) ET
- sous réserve que l'on n'ait pas des relations contradictoires (exemple : ADA induction ADA vs. ADA repression ADA). On ne devrait avoir au final que des régulations de type +/induction, -/repression, +-/complex, ?/unknown
Script pour le filtrage :
Utilisation : perl filter_interactions.pl Ecoli.GPL3154.rma.regulondb.txt network_tf_gene.unfiltered.txt > network_tf_gene.filtered.txt
Inférence du réseau de régulation
Nous allons utiliser maintenant la librairie minet (Meyer PE, Lafitte F, Bontempi G., 2008) pour reconstruire le réseau. Une version plus complète de la vignette (nom de la documentation/tutorial en R) se trouve ici.
Cette librairie résultant d'une étude sur la construction d'un estimateur de l'information mutuelle entre 2 variables, la documentation est très axée sur les estimateurs proposés. Pour aujourd'hui, chacun d'entre vous pourra prendre un estimateur différent (y compris la matrice de corrélation) afin de comparer les résultats obtenus.
Suivez-donc le tutoriel, en adaptant les commandes aux jeux de données préalablement constitués jusqu'à présent. Les étapes sont les suivantes :
- chargement des données d'expression
- discrétisation (optionnel pour la matrice de corrélation) (plusieurs méthodes possibles)
- construction de la matrice de corrélation ou d'information mutuelle (plusieurs méthodes ou estimateurs disponibles)
- inférence du réseau (seuil à déterminer pour prédire la présence d'un(e) arc/arête)
A ce stade, vous aurez besoin du réseau de référence pour évaluer la qualité de la reconstruction. La fonction validate de la librairie prend en paramètre une matrice d'adjacence (pour la représentation du réseau de référence). Il faut donc convertir le fichier SIF en matrice d'adjacence.
Utilisez le script sif-2-adjency-matrix.pl pour convertir le fichier SIF et le charger dans R. Puis, utilisez la fonction validate pour comparer le réseau inféré au réseau de référence. La procédure balaye le seuil sur "le niveau de confiance" minimum pour inférer une régulation.
D'après le résultat de la fonction validate, quel est le seuil pour une précision de 60% ? 30% ?
A ce stade, il est possible de sauvegarder le réseau pour le visualiser avec Cytoscape (ou autre). Le résultat des fonctions d'extraction du réseau (clr, aracne, ...) n'est autre qu'une matrice d'adjacence contenant des valeurs de confiance dans la présence d'un arc.
Cependant, comme l'ont fait Faith et ses collègues, il apparaît judicieux d'orienter le graphe et de n'autoriser les arcs à ne sortir que de régulateurs (ou de gènes codant pour des protéines arborant un domaine de liaison l'ADN). Pour cela, il suffit de charger le fichier SIF et de mettre à 0 les arcs qui ne sortent pas de facteur de transcription :
sif=read.table('Ecoli.grn.sif', header=F) # chargement notTF=setdiff(sif$V3,sif$V1) # label des sommets qui ne sont pas des TFs net2=net # recopie de la matrice d'ajacence net2[notTF,]=0 # mise à zéro des arcs sortants pour ces sommets
Sauvegardez le réseau et convertissez-le au format tabulé de Cytoscape pour le visualiser. Pour cela, déterminez le seuil de confiance que vous souhaitez utiliser et modifier le script suivant en conséquence adjency-matrix-2-cytoscape-tab.pl ; son utilisation est la suivante :
./adjency-matrix-2-cytoscape-tab.pl EcolA.inferred.grn.mat > EcolA.inferred.grn.tab
Pour allez plus loin...
Essayez la méthode mise en oeuvre par Banjo pour reconstruire des réseaux bayésiens statiques et/ou dynamiques.



