M1 MABS Graphes TP Dessin et Introduction iGraph
From silico.biotoul.fr
(Difference between revisions)
m (Created page with '= Dessin de graphes = Dans cette partie, nous allons voir comment lire/écrire un arbre au format Newick. Puis, comment afficher l'arborescence avec différents algorithmes : * …') |
m |
||
| Line 34: | Line 34: | ||
def lastChild(node): return node['children'][ len(node['children'])-1 ] | def lastChild(node): return node['children'][ len(node['children'])-1 ] | ||
| - | def parseNewick(text): | + | def parseNewick(text): # ROUGH Newick PARSING: no syntax check, unsecure, fault intolerent!!! |
T = createTree() | T = createTree() | ||
i=0 | i=0 | ||
Revision as of 10:48, 14 March 2012
Dessin de graphes
Dans cette partie, nous allons voir comment lire/écrire un arbre au format Newick. Puis, comment afficher l'arborescence avec différents algorithmes :
- textuelle comme par exemple une arborescence de fichiers
- graphique

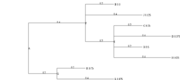
Un arbre phylogénétique récupéré sur le site PATRIC. Télécharger au format Newick.
Pour cela, nous allons utiliser les arbres ci-contre.
Chargement d'un arbre au format Newick
La première étape consiste donc à pouvoir charger l'arbre au format Newick dans un script python.
Pour cela, il semble opportun de se créer une petite bibliothèque pour manipuler les arbres : Tree.py
def createTree(): T = {} T['root'] = None return T def createTreeNode(): N = { 'parent': None, 'children': [], 'label': '', 'distance': 1 } return N def isLeaf(node): return len(node['children'])==0 def firstChild(node): return node['children'][0] def lastChild(node): return node['children'][ len(node['children'])-1 ] def parseNewick(text): # ROUGH Newick PARSING: no syntax check, unsecure, fault intolerent!!! T = createTree() i=0 stack=[] current=createTreeNode() # root while i < len(text): if text[i]==' ': # skip spaces i+=1 if text[i]==';': # finished ! i+=1 elif text[i]=='(': # new child N=createTreeNode() current['children'].append(N) N['parent'] = current stack.insert(0,current) current = N i+=1 elif text[i]==',': # end of label or branch length... next child incoming N=createTreeNode() parent = stack[0] # parent is at the top of the stack parent['children'].append(N) N['parent'] = parent current = N i+=1 elif text[i]==')': # new child complete current=stack.pop(0) i+=1 elif text[i]==':': # incoming branch length # parse number nbText = '' i+=1 while text[i] in ['0','1','2','3','4','5','6','7','8','9','0','.','E','e','-']: nbText+=text[i] i+=1 current['distance'] = float(nbText) else: # maybe incoming node label label='' while not text[i] in [' ',')',':',',',';']: label+=text[i] i+=1 current['label'] = label return current def loadNewick(filename): with open(filename) as f: text = f.readline().rstrip() T = parseNewick(text) return T