M1 MABS Graphes TP Dessin et Introduction iGraph
From silico.biotoul.fr
Contents |
Dessin de graphes

Dans cette partie, nous allons voir comment lire/écrire un arbre au format Newick. Puis, comment afficher l'arborescence avec différents algorithmes :
- textuelle comme par exemple une arborescence de fichiers
- graphique
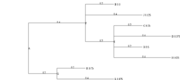
Pour cela, nous allons utiliser les arbres ci-contre.
Chargement d'un arbre au format Newick
La première étape consiste donc à pouvoir charger l'arbre au format Newick dans un script python.
Pour cela, il semble opportun de se créer une petite bibliothèque pour manipuler les arbres : Tree.py
def createTree(): return { 'root': None } def createTreeNode(): return { 'parent': None, 'children': [], 'label': '', 'distance': 1 } def isLeaf(node): return len(node['children'])==0 def firstChild(node): return node['children'][0] def lastChild(node): return node['children'][ len(node['children'])-1 ] # ROUGH Newick PARSING: ## no syntax checking, ## unsecure, ## absolutely not fault tolerant/robust, ## not guaranteed to parse any newick compliant file!!! def parseNewick(text): T = createTree() i=0 stack=[] current=createTreeNode() # root while i < len(text): if text[i]==' ': # skip spaces i+=1 if text[i]==';': # finished ! i+=1 elif text[i]=='(': # new child N=createTreeNode() current['children'].append(N) N['parent'] = current stack.insert(0,current) current = N i+=1 elif text[i]==',': # end of label or branch length... next child incoming N=createTreeNode() parent = stack[0] # parent is at the top of the stack parent['children'].append(N) N['parent'] = parent current = N i+=1 elif text[i]==')': # new child complete current=stack.pop(0) i+=1 elif text[i]==':': # incoming branch length # parse number nbText = '' i+=1 while text[i] in ['0','1','2','3','4','5','6','7','8','9','0','.','E','e','-']: nbText+=text[i] i+=1 current['distance'] = float(nbText) else: # maybe incoming node label label='' while not text[i] in [' ',')',':',',',';']: label+=text[i] i+=1 current['label'] = label return current def loadNewick(filename): with open(filename) as f: text = f.readline().rstrip() T = parseNewick(text) return T
Affichage au format texte
Écrire une procédure pour afficher un arbre sous forme d'arborescence de fichier. Le résultat pour toyExample devrait ressembler à ce qui suit :
|-0.2- A | |-0.4- F | | |-0.2- B | | |-0.4- J | | |-0.2- E | | | |-0.2- C | | | |-0.4- D | | | |-0.2- H | | | |-0.4- I | |-0.2- G | | |-0.2- K | | |-0.4- L
Écrire une procédure pour exporter l'arbre au format Newick.
Dessin de l'arborescence sous forme non circulaire
Pour cela, nous allons utiliser une astuce qui consiste à générer du texte au format SVG (du xml) pour visualiser le résultat avec Firefox ou un autre logiciel capable d'afficher ce format. Il s'agit donc de placer les étiquettes sur un canevas ainsi que de tracer des lignes correspondant aux branches.
Pour vous faire une idée du format SVG, consulter le contenu du fichier SVG généré pour toyExample.
Principe :
- Calculer la hauteur et le nombre de feuilles. Ceci va servir à normaliser entre 0 et 1 les coordonnées (x,y) de chaque sommet et ainsi on pourra afficher sur un canevas de taille (largeur,hauteur) en multipliant : x*largeur et y*hauteur pour avoir les coordonnées dans cette taille d'image.
- Pour les coordonnées sur l'axe des Y :
- répartir les feuilles uniformément (entre 0 et 1)
- à l'aide d'une procédure récursive, une fois les coordonnées Y des descendants connues, la coordonnées Y du sommet est entre le premier et le dernier descendant.
- Pour les coordonnées sur l'axe des X :
- Calculer la profondeur ou distance de chaque sommet à la racine (normalisée entre 0 et 1)
Utilisation de la bibliothèque iGraph sous R
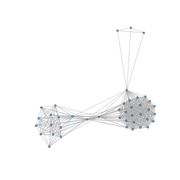
Pour cela nous allons travailler avec le graphe ci-contre dans lequel les sommets correspondent à des gènes de différents organismes procaryotes et les liens correspondent à une relation d'isorthologie inférée entre les gènes.