M1 MABS Graphes TP Parcours en largeur - Plus court chemin
From silico.biotoul.fr
(Difference between revisions)
m (→Plus court chemin - Dijkstra) |
m (→Parcours en largeur) |
||
| Line 1: | Line 1: | ||
= Parcours en largeur = | = Parcours en largeur = | ||
Algorithme : | Algorithme : | ||
| - | BFS( | + | BFS(G, s) |
| - | + | for each vertex u in V(G) | |
| - | + | do color[u] <- white | |
| - | + | d[u] <- inf | |
| - | + | pred[u] <- NIL | |
| - | + | color[s] <- gray | |
| - | + | d[s] <- 0 | |
| - | + | Q <- <math>\empty</math> | |
| - | + | enqueue(Q, s) | |
| - | + | while Q <math>\ne</math> <math>\empty</math> | |
| - | + | do u <- dequeue(Q) | |
| + | for each v in Adj[u] | ||
| + | do if color[v] = white | ||
| + | then color[v] <- gray | ||
| + | d[v] <- d[u] + 1 | ||
| + | pred[v] <- u | ||
| + | enqueue(Q, v) | ||
| + | color[u] <- black | ||
| - | Charger le graphe de la séance précédente, et faire un parcours en largeur depuis | + | Charger le graphe de la séance précédente ([[File:M1MABS Graphe dressing.sif]]), et faire un parcours en largeur depuis sous-vetements. |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
= Plus court chemin - Dijkstra = | = Plus court chemin - Dijkstra = | ||
Revision as of 11:52, 4 February 2015
Contents |
Parcours en largeur
Algorithme :
BFS(G, s)
for each vertex u in V(G)
do color[u] <- white
d[u] <- inf
pred[u] <- NIL
color[s] <- gray
d[s] <- 0
Q <- 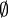 enqueue(Q, s)
while Q
enqueue(Q, s)
while Q 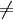
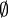 do u <- dequeue(Q)
for each v in Adj[u]
do if color[v] = white
then color[v] <- gray
d[v] <- d[u] + 1
pred[v] <- u
enqueue(Q, v)
color[u] <- black
do u <- dequeue(Q)
for each v in Adj[u]
do if color[v] = white
then color[v] <- gray
d[v] <- d[u] + 1
pred[v] <- u
enqueue(Q, v)
color[u] <- black
Charger le graphe de la séance précédente (File:M1MABS Graphe dressing.sif), et faire un parcours en largeur depuis sous-vetements.
Plus court chemin - Dijkstra
Pour l'algorithme suivant,
- D[x] donne la plus courte distance entre s et x. Au départ, D[s]=0 et D[x]=infini pour tout x différent s.
- P[x] est le prédécesseur de x dans le chemin de plus courte distance allant de s à x. Au départ, P[x]=rien pour tout x.
- C est l'ensemble de sommets qu'il reste à parcourir. Au départ, C=V (V étant l'ensemble des sommets du graphe).
- W[x,y] est la valuation de l'arc (x,y)
Algorithme :
Dijkstra(graphe g, sommet s)
initialiser D, P et C
tant que C n'est pas vide faire
x = extraire le sommet le plus proche de C
pour chaque successeur de x faire
si D[x] + W[x,successeur] < D[successeur] alors
D[successeur] = D[x] + W[successeur]
P[successeur] = x
Extraire la composante connexe contenant WBBJ, puis faire tourner l'algorithme ci-dessus à partir de WBBJ. Pour la valuation des arêtes, on prendra 1000-combined.
Résultat attendu :
{'distances':
{'WBBI': 29,
'WBBH': 34,
'RFBX': 71,
'WBBJ': 0,
'RFBC': 103,
'RFBD': 104,
'WBBK': 6},
'predecessors': {
'WBBI': 'WBBK',
'WBBH': 'WBBK',
'RFBX': 'WBBJ',
'RFBC': 'RFBX',
'RFBD': 'RFBC',
'WBBK': 'WBBJ'}}
Plus courts chemins - Floyd-Warshall
Pour l'agorithme suivant,
- D[x,y] est la distance du plus court chemin entre les sommets x et y
- N[x,y] est le successeur de x dans le plus court chemin allant de x à y
- W[x,y] est la valuation de l'arc (x,y)
Algorithme :
initialiser D = W, et N
pour k de 1 à n
pour i de 1 à n
pour j de 1 à n
si D[i,k] + D[k,j] < D[i,j] alors
D[i,j] = D[i,k] + D[k,j]
N[i,j] = k
Récupération du plus court chemin à partir de la matrice N :
plusCourtChemin(D,N, i,j)
si D[i,j] est infinie alors
il n'y a pas de chemin entre i et j
chemin = initialiserChemin(i)
k = N[i,j]
tant que k est défini faire
ajouter(chemin, k)
k = N[k,j]
ajouter(chemin, j)
Implémenter les algorithmes ci-dessus en R et les tester sur la matrice suivante :
# adjacency matrix M=matrix( c(0,1,0,0,1,0,0,1,0,0,1,1,0,0,0,0), ncol=4, byrow=T) M # [,1] [,2] [,3] [,4] #[1,] 0 1 0 0 #[2,] 1 0 0 1 #[3,] 0 0 1 1 #[4,] 0 0 0 0 # test fw=FloydWarshall(M) fw #$distances # [,1] [,2] [,3] [,4] #[1,] 2 1 Inf 2 #[2,] 1 2 Inf 1 #[3,] Inf Inf 1 1 #[4,] Inf Inf Inf Inf # #$chemins # [,1] [,2] [,3] [,4] #[1,] 2 NA Inf 2 #[2,] NA 1 Inf NA #[3,] Inf Inf NA NA #[4,] Inf Inf Inf Inf FloydWarshallPath(fw, 1,4) #[1] 1 2 4
Extraire la grosse composante connexe de String au format sif puis transformer le fichier en matrice d'adjacence. Ensuite, sous R, charger la matrice et calculer le diamètre du graphe.



