M1 MABS Graphes TP Parcours en largeur - Plus court chemin
From silico.biotoul.fr
m (→Plus courts chemins - Floyd-Warshall) |
m (→Plus courts chemins - Floyd-Warshall) |
||
| (16 intermediate revisions not shown) | |||
| Line 1: | Line 1: | ||
| - | = Parcours en | + | = Parcours en largeur = |
Algorithme : | Algorithme : | ||
| - | BFS( | + | BFS(G, s) |
| - | + | '''for each''' vertex u <math>\in</math> V(G) | |
| - | + | '''do''' color[u] <math>\leftarrow</math> WHITE | |
| - | + | d[u] <math>\leftarrow</math> <math>\infty</math> | |
| - | + | <math>\pi</math>[u] <math>\leftarrow</math> NIL | |
| - | + | color[s] <math>\leftarrow</math> GRAY | |
| - | + | d[s] <math>\leftarrow</math> 0 | |
| - | + | Q <math>\leftarrow</math> <math>\empty</math> | |
| - | + | enqueue(Q, s) | |
| - | + | '''while''' Q <math>\ne</math> <math>\empty</math> | |
| - | + | '''do''' u <math>\leftarrow</math> dequeue(Q) | |
| + | '''for each''' vertex v <math>\in</math> Adj[u] | ||
| + | '''do if''' color[v] = WHITE | ||
| + | '''then''' color[v] <math>\leftarrow</math> GRAY | ||
| + | d[v] <math>\leftarrow</math> d[u] + 1 | ||
| + | <math>\pi</math>[v] <math>\leftarrow</math> u | ||
| + | enqueue(Q, v) | ||
| + | color[u] <math>\leftarrow</math> BLACK | ||
| - | Charger le graphe de la séance précédente, et | + | '''Charger''' le graphe de la séance précédente ([[File:M1MABS Graphe dressing.sif]]), et effectuer un parcours en largeur depuis sous-vetements. |
| - | + | = Plus court chemin - Bellman-Ford = | |
| - | + | Rappel de l'algorithme: | |
| - | + | INITIALIZE-SINGLE-SOURCE(G, s) | |
| - | + | '''for each''' vertex v <math>\in</math> V(G) | |
| - | + | '''do''' d[v] <math>\leftarrow</math> <math>\infty</math> | |
| + | <math>\pi</math>[v] <math>\leftarrow</math> NIL | ||
| + | d[s] <math>\leftarrow</math> 0 | ||
| + | RELAX(u, v, w) | ||
| + | '''if''' d[v] > d[u] + w(u, v) | ||
| + | '''then''' d[v] = d[u] + w(u,v) | ||
| + | <math>\pi</math>[v] <math>\leftarrow</math> u | ||
| + | BELLMAN-FORD(G, s, w) | ||
| + | INITIALIZE-SINGLE-SOURCE(G, s) | ||
| + | '''for''' i <math>\leftarrow</math> 1 '''to''' |V(G)| - 1 | ||
| + | '''do for each''' edge (u,v) <math>\in</math> E(G) | ||
| + | '''do''' RELAX(u, v, w) | ||
| + | |||
| + | '''Ajoutez''' la méthode à votre librairie et testez la sur le graphe [[File:M1MABS Graphe Bellman-Ford.tab]] à partir du sommet A. | ||
| + | |||
| + | Afin de gagner du temps, la méthode <tt>loadTAB</tt> vous est fournie : | ||
| + | <source lang='python'> | ||
| + | def loadTAB(self, filename): | ||
| + | """ | ||
| + | Loads a graph in Cytoscape tab format | ||
| + | Assumed input: | ||
| + | id1 id2 weight color ... | ||
| + | A B 6 blue ... | ||
| + | """ | ||
| + | with open(filename) as f: | ||
| + | # GET COLUMNS NAMES | ||
| + | tmp = f.readline().rstrip() | ||
| + | attNames= tmp.split('\t') | ||
| + | # REMOVES FIRST TWO COLUMNS WHICH CORRESPONDS TO THE LABELS OF THE CONNECTED VERTICES | ||
| + | attNames.pop(0) | ||
| + | attNames.pop(0) | ||
| + | # PROCESS THE REMAINING LINES | ||
| + | row = f.readline().rstrip() | ||
| + | while row: | ||
| + | vals = row.split('\t') | ||
| + | v1 = vals.pop(0) | ||
| + | v2 = vals.pop(0) | ||
| + | att = {} | ||
| + | for i in xrange(len(attNames)): | ||
| + | att[ attNames[i] ] = vals[i] | ||
| + | self.addEdge(v1, v2, att) | ||
| + | row = f.readline().rstrip() # NEXT LINE | ||
| + | </source> | ||
| + | <!-- | ||
= Plus court chemin - Dijkstra = | = Plus court chemin - Dijkstra = | ||
Pour l'algorithme suivant, | Pour l'algorithme suivant, | ||
| Line 35: | Line 85: | ||
pour chaque successeur de x faire | pour chaque successeur de x faire | ||
si D[x] + W[x,successeur] < D[successeur] alors | si D[x] + W[x,successeur] < D[successeur] alors | ||
| - | D[successeur] = D[x] + | + | D[successeur] = D[x] + W[successeur] |
P[successeur] = x | P[successeur] = x | ||
| Line 56: | Line 106: | ||
'RFBD': 'RFBC', | 'RFBD': 'RFBC', | ||
'WBBK': 'WBBJ'}} | 'WBBK': 'WBBJ'}} | ||
| + | --> | ||
= Plus courts chemins - Floyd-Warshall = | = Plus courts chemins - Floyd-Warshall = | ||
| Line 64: | Line 115: | ||
Algorithme : | Algorithme : | ||
| - | initialiser D = W, et N | + | initialiser D = W, et N à partir des arcs/arêtes du graphe |
pour k de 1 à n | pour k de 1 à n | ||
pour i de 1 à n | pour i de 1 à n | ||
| Line 70: | Line 121: | ||
si D[i,k] + D[k,j] < D[i,j] alors | si D[i,k] + D[k,j] < D[i,j] alors | ||
D[i,j] = D[i,k] + D[k,j] | D[i,j] = D[i,k] + D[k,j] | ||
| - | N[i,j] = k | + | N[i,j] = N[i,k] |
Récupération du plus court chemin à partir de la matrice N : | Récupération du plus court chemin à partir de la matrice N : | ||
| Line 82: | Line 133: | ||
k = N[k,j] | k = N[k,j] | ||
ajouter(chemin, j) | ajouter(chemin, j) | ||
| + | |||
| + | '''Implémentez''' les méthodes FloydWarshall(W) et FloydWarshallPath(source, destination) dans votre librairie. Pour cela, vous êtes forcement incité(e) à ajouter une méthode ''adjacencyMatrix'' qui renvoie le graphe source forme de matrice d'adjacence. | ||
| + | |||
| + | |||
| + | '''Testez''' ces méthodes sur le graphe [[File:M1MABS Graphe Floyd-Warshall.tab]] et '''affichez''' les plus courts chemins de A à C et de A à B (avec leur longueur). | ||
| + | |||
| + | Quel est le diamètre du graphe ? '''Affichez''' le chemin correspondant. | ||
| + | |||
| + | |||
| + | <!-- | ||
| + | <source lang='rsplus'> | ||
| + | # adjacency matrix | ||
| + | M=matrix( c(0,1,0,0,1,0,0,1,0,0,1,1,0,0,0,0), ncol=4, byrow=T) | ||
| + | M | ||
| + | # [,1] [,2] [,3] [,4] | ||
| + | #[1,] 0 1 0 0 | ||
| + | #[2,] 1 0 0 1 | ||
| + | #[3,] 0 0 1 1 | ||
| + | #[4,] 0 0 0 0 | ||
| + | |||
| + | # test | ||
| + | fw=FloydWarshall(M) | ||
| + | fw | ||
| + | #$distances | ||
| + | # [,1] [,2] [,3] [,4] | ||
| + | #[1,] 2 1 Inf 2 | ||
| + | #[2,] 1 2 Inf 1 | ||
| + | #[3,] Inf Inf 1 1 | ||
| + | #[4,] Inf Inf Inf Inf | ||
| + | # | ||
| + | #$chemins | ||
| + | # [,1] [,2] [,3] [,4] | ||
| + | #[1,] 2 NA Inf 2 | ||
| + | #[2,] NA 1 Inf NA | ||
| + | #[3,] Inf Inf NA NA | ||
| + | #[4,] Inf Inf Inf Inf | ||
| + | |||
| + | FloydWarshallPath(fw, 1,4) | ||
| + | #[1] 1 2 4 | ||
| + | </source> | ||
| + | |||
Extraire la grosse composante connexe de String au format sif puis transformer le fichier en matrice d'adjacence. Ensuite, sous R, charger la matrice et calculer le diamètre du graphe. | Extraire la grosse composante connexe de String au format sif puis transformer le fichier en matrice d'adjacence. Ensuite, sous R, charger la matrice et calculer le diamètre du graphe. | ||
| + | |||
| + | = Données = | ||
| + | * [[Media:String_EcolA_selection.tab|String_EcolA_selection.tab]] | ||
| + | * [[Media:WBBJ_EcolA_String_subgraph.tab|WBBJ_EcolA_String_subgraph.tab]] | ||
| + | --> | ||
Current revision as of 15:22, 20 February 2015
Parcours en largeur
Algorithme :
BFS(G, s) for each vertex uV(G) do color[u]
WHITE d[u]
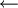
π[u]
NIL color[s]
GRAY d[s]
0 Q
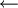
enqueue(Q, s) while Q
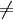
do u
dequeue(Q) for each vertex v
Adj[u] do if color[v] = WHITE then color[v]
GRAY d[v]
d[u] + 1 π[v]
u enqueue(Q, v) color[u]
BLACK
Charger le graphe de la séance précédente (File:M1MABS Graphe dressing.sif), et effectuer un parcours en largeur depuis sous-vetements.
Plus court chemin - Bellman-Ford
Rappel de l'algorithme:
INITIALIZE-SINGLE-SOURCE(G, s) for each vertex vV(G) do d[v]
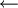
π[v]
NIL d[s]
0 RELAX(u, v, w) if d[v] > d[u] + w(u, v) then d[v] = d[u] + w(u,v) π[v]
u BELLMAN-FORD(G, s, w) INITIALIZE-SINGLE-SOURCE(G, s) for i
1 to |V(G)| - 1 do for each edge (u,v)
E(G) do RELAX(u, v, w)
Ajoutez la méthode à votre librairie et testez la sur le graphe File:M1MABS Graphe Bellman-Ford.tab à partir du sommet A.
Afin de gagner du temps, la méthode loadTAB vous est fournie :
def loadTAB(self, filename): """ Loads a graph in Cytoscape tab format Assumed input: id1 id2 weight color ... A B 6 blue ... """ with open(filename) as f: # GET COLUMNS NAMES tmp = f.readline().rstrip() attNames= tmp.split('\t') # REMOVES FIRST TWO COLUMNS WHICH CORRESPONDS TO THE LABELS OF THE CONNECTED VERTICES attNames.pop(0) attNames.pop(0) # PROCESS THE REMAINING LINES row = f.readline().rstrip() while row: vals = row.split('\t') v1 = vals.pop(0) v2 = vals.pop(0) att = {} for i in xrange(len(attNames)): att[ attNames[i] ] = vals[i] self.addEdge(v1, v2, att) row = f.readline().rstrip() # NEXT LINE
Plus courts chemins - Floyd-Warshall
Pour l'agorithme suivant,
- D[x,y] est la distance du plus court chemin entre les sommets x et y
- N[x,y] est le successeur de x dans le plus court chemin allant de x à y
- W[x,y] est la valuation de l'arc (x,y)
Algorithme :
initialiser D = W, et N à partir des arcs/arêtes du graphe
pour k de 1 à n
pour i de 1 à n
pour j de 1 à n
si D[i,k] + D[k,j] < D[i,j] alors
D[i,j] = D[i,k] + D[k,j]
N[i,j] = N[i,k]
Récupération du plus court chemin à partir de la matrice N :
plusCourtChemin(D,N, i,j)
si D[i,j] est infinie alors
il n'y a pas de chemin entre i et j
chemin = initialiserChemin(i)
k = N[i,j]
tant que k est défini faire
ajouter(chemin, k)
k = N[k,j]
ajouter(chemin, j)
Implémentez les méthodes FloydWarshall(W) et FloydWarshallPath(source, destination) dans votre librairie. Pour cela, vous êtes forcement incité(e) à ajouter une méthode adjacencyMatrix qui renvoie le graphe source forme de matrice d'adjacence.
Testez ces méthodes sur le graphe File:M1MABS Graphe Floyd-Warshall.tab et affichez les plus courts chemins de A à C et de A à B (avec leur longueur).
Quel est le diamètre du graphe ? Affichez le chemin correspondant.



