Examen terminal
From silico.biotoul.fr
Contents |
Instructions pour l'examen, Durée 2h
Vous donnerez les réponses aux questions sur la feuille d'examen fournie. Vous pouvez utiliser toutes les ressources informatiques nécessaires, les principaux liens vers les sites sont présentés ci-dessous.
Ressources
Vous trouverez ci-dessous une sélection des sites Internet qui vous seront nécessaires pour l'examen:
- Génopôle Toulouse
- EBI European Bioinformatics Institute (EMBL, GB)
- NCBI National Center for Biotechnology Information (NIH, USA)
- Expasy Expert Protein Analysis System (Swiss Institute of Bioinformatics, Suisse)
- PBIL Pôle Bio-Informatique Lyonnais (CNRS, Lyon)
- Institut Pasteur
- Protein Data Bank (PDB)
- EMBOSS.
Exercice 1
- 1.1 Utiliser la méthode de programmation dynamique pour déterminer l’alignement global optimal entre les deux séquences suivantes :
Séquence 1 : GTCCATG
Séquence 2 : CCAC
Système de scores : identité = 0, substitution = +1, indel = +2 (Utilisation pour le calcul d’un score de distance)
Remplir la matrice de programmation dynamique et produire l’alignement final. Quel est le score de cet alignement ?
- 1.2 Expliquer pourquoi la pénalité des indels doit être plus importante que la pénalité des substitutions.
Exercice 2
Parmi les questions suivantes, indiquez sur votre copie, lesquelles sont VRAIES:
- 1. Blast est un programme d’alignement global
- 2. Blast calcule une E-value : une valeur comprise entre 0 et 1
- 3. Blast calcule une E-value reflétant le nombre attendu d’alignements significatifs de score supérieur ou égal à celui obtenu entre la séquence requête et la séquence de la banque.
- 4. La programmation dynamique est une méthode utilisée uniquement pour réaliser un alignement global entre 2 séquences
- 5. Le numéro d’accession d’une séquence est définitif.
- 6. Le pourcentage de similarité est toujours supérieur ou égal au pourcentage d’identité
- 7. PUBMED est une méthode pour prédire les sites actifs d’une protéine
- 8. G-A-[ILV]-X-D est une signature PROSITE
- 9. Un motif protéique ne peut pas être présent dans un domaine fonctionnel
- 10. ENTREZ permet d'identifier des structures protéiques
- 11. Il existe des banques de données dites de connaissances
- 12. Le dot-plot permet de visualiser les régions répétées présentes dans une séquence
Exercice 3
L'équipe dans laquelle vous travailler vient d’obtenir la séquence d’un ADNc d’intérêt majeur pour son travail de recherche. Vous devez identifiez la protéine codée correspondante et en rélaiser la prédiction fonctionnelle (fonction putative, famille protéique, motifs...)
- 3.1 Justifiez chaque étape de votre travail et expliquez les hypothèses faites
Exercice 4
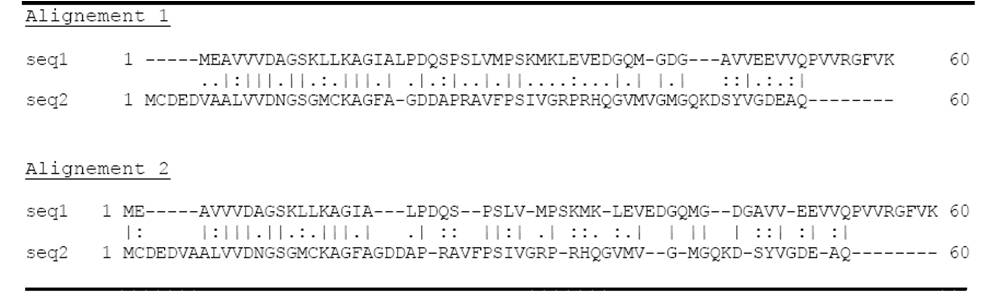
Les 60 premiers acides aminés de deux protéines ont été alignés en utilisant un alignement global mais en faisant varier des paramètres.
- 3.1. Entre ces deux alignements quel(s) paramètre(s) a-t-on fait varier ?
- 3.2. De quelle manière ?
- 3.3. Du point de vue biologique, quel est, à votre avis, le meilleur alignement? Justifiez votre réponse.
Exercice 5
Recherchez la séquence ABL11548 dans les bases de données, et répondez aux questions ci-dessous.
- 4.1 Quel est le nom de la protéine ?
- 4.2 Quelle est la taille de cette protéine? Est-elle complète ? Expliquez.
- 4.3 Combien de domaines contient cette protéine ? Donnez en le(s) nom(s) et position(s)
Recherchez la séquence CAA65843 dans les bases de données, et répondez aux questions ci-dessous.
- 4.4 Faites une représentation schématique des deux protéines où vous ferez apparaître les domaines, et les zones alignées.
- 4.5 Que pouvez vous conclure par rapport à l’annotation de la séquence ABL11548 ?
Ci-dessous est présenté l’alignement multiple représentatif du domaine CBM_1 tel qu’il est présenté dans la banque de domaines SMART
- 4.6 A partir de cet alignement multiple, dégager une signature PROSITE
- 4.7 Donnez la/les position(s) du/des motif(s) CBM_1 sur la séquence ABL11548 et CAA65843