InfoBio TD Matrices PAM
From silico.biotoul.fr
Manipulation de matrices avec R
A = matrix( c(11, 12, 13, 21, 22, 23, 31, 32, 33), ncol=3, byrow=TRUE )
Cas de la matrice unité d'ordre n (notée In), exemple avec n = 3 :
I3 = diag(3)
Cas des vecteurs colonne ou ligne :
V = c(1, 0, 5) class(V) # obtention de la matrice vecteur colonne : as.matrix(V) # pour obtenir la matrice vecteur ligne, on fait la transposée du résultat pécédent : t( as.matrix(V) )
- addition +, soustraction -, multiplication par un nombre *,
- produit matriciel :
- entre 2 matrices A et I3 : A %*% I3,
- entre une matrice A et un vecteur v : A %*% v
- transposition : t(M)
- trace : sum( diag(M) )
- déterminant : det(M)
- inversion d'une matrice : solve(M)
- calcul des valeurs propres et des vecteurs propres : eigen(M)
Application aux matrices de substitution pour les alignements de séquences protéiques
cf. Alignement de deux séquences protéiques concernant le cours.
Nous allons construire une matrice de type Dayhoff (PAM) pour l'alignement de séquences protéiques, ceci à partir d'alignements de séquences. La méthode en quelques mots consiste à
- compter le nombre de substitutions observées pour chaque acide aminé (a.a.),
- à transformer ce nombre d'occurrence en fréquence,
- ramener les fréquences de mutations telles que l'on ait 1 mutation acceptée sur 100 a.a. Cela correspond à une unité de distance évolutive (PAM1).
- multiplier la matrice par elle-même pour obtenir la distance évolutive souhaitée (ici PAM120)
- calculer la log odds matrix : la matrice contenant le coût attribué à chaque substitution (match et mismatch).
Fréquence de mutation d'un a.a. en un autre à partir d'alignements
La première étape (déjà réalisée pour vous) consiste à réaliser les alignements et à dénombrer le nombre de passage d'un acide aminé à un autre.
Fichier contenant, le nombre de substitutions observées entre chaque paire d'acides aminés dans un lot de séquences préalablement alignées File:Count matrix.txt.
CM=read.table("count_matrix.txt", sep="\t", header=TRUE, row.names=1)
Après avoir chargé cette matrice dans R, transformez-la en une matrice MM (mutation matrix) qui contient des fréquences (aidez-vous des fonctions apply et sum). Ces fréquences sont interprétées comme des probabilités de passer d'un acide aminé en un autre.
Vous devriez obtenir ceci (10 premières lignes et 10 premières colonne) :
round(MM[1:10,1:10],2) A R N D C Q E G H I A 0.54 0.03 0.03 0.03 0.08 0.04 0.05 0.05 0.02 0.02 R 0.02 0.57 0.03 0.01 0.02 0.06 0.03 0.01 0.04 0.01 N 0.01 0.02 0.48 0.06 0.01 0.03 0.03 0.03 0.05 0.01 D 0.02 0.01 0.09 0.57 0.01 0.04 0.11 0.02 0.03 0.00 C 0.01 0.00 0.00 0.00 0.57 0.00 0.00 0.00 0.00 0.00 Q 0.02 0.04 0.03 0.02 0.01 0.42 0.05 0.01 0.04 0.01 E 0.04 0.04 0.05 0.13 0.01 0.11 0.52 0.02 0.03 0.01 G 0.05 0.02 0.05 0.03 0.02 0.02 0.02 0.77 0.02 0.00 H 0.00 0.01 0.02 0.01 0.01 0.02 0.01 0.00 0.54 0.00 I 0.02 0.01 0.01 0.00 0.03 0.01 0.01 0.00 0.01 0.50
Fréquences des a.a. dans les séquences de départ
Nous avons donc besoin de calculer la fréquences de chaque acide aminé dans une séquence (ici, fréquences d'apparition dans nos alignements de départ). Utilisez donc la matrice CM de départ qui contient le nombre de substitutions observées pour calculer le vecteur Freq contenant la fréquence de chacun de a.a.
Vous devriez obtenir le vecteur suivant :
round(Freq,2) A R N D C Q E G H I L K M F P S T W Y V 0.08 0.05 0.04 0.06 0.01 0.03 0.07 0.09 0.02 0.07 0.09 0.06 0.02 0.04 0.04 0.05 0.06 0.01 0.03 0.08
Taux de mutations correspondant à la matrice MM
A partir des fréquences d'apparition des a.a. calculées ci-dessus et de leur probabilité de muter, calculez le nombre de mutations que l'on s'attend à avoir sur une séquence de 100 a.a. Vous devriez obtenir 43,9 mutations.
Recherche de MM1 telle que le taux de mutation soit 0.01
La matrice PAM1 correspond à 1 mutation acceptée sur 100 a.a., le taux de mutation de cette matrice doit donc être 0.01 or ce n’est pas le cas de la matrice MM.
Le pourcentage d’a.a. qui ne change pas est
| x = 100 | ∑ | fiMii |
| i |
avec Mii la probabilité de ne pas muter ou bien encore
| x = 100 | ∑ | fi(1 − λmi) |
| i |
avec mi la probabilité de muter.
on en déduit 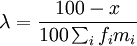 et on peut mettre à l'échelle MM afin que le pourcentage de mutations soit 1 :
et on peut mettre à l'échelle MM afin que le pourcentage de mutations soit 1 : 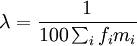 .
.
On a donc 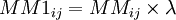 si i différent j et sinon
si i différent j et sinon 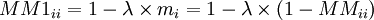
Vous devriez obtenir ceci (10 premières lignes et 10 premières colonne) :
round(MM1*10000)[1:10,1:10] A R N D C Q E G H I A 9897 6 7 7 18 10 11 12 5 5 R 3 9902 6 3 4 13 6 3 8 2 N 3 5 9881 13 3 6 6 6 10 1 D 5 3 20 9902 2 9 26 5 8 1 C 3 1 1 0 9903 0 0 1 1 1 Q 4 9 6 5 1 9867 12 2 8 1 E 9 8 10 31 1 24 9890 4 7 1 G 11 4 12 6 4 4 5 9948 3 1 H 1 3 6 3 1 5 2 1 9895 0 I 4 2 2 1 6 2 1 1 2 9886
Calcul de la matrice de substitution
Pour un alignement de l'acide aminé AAi avec l'acide aminé AAj, on utilise la formule suivante :

avec M la matrice MM précédemment calculée (mutation matrix) et F la fréquence de chacun des acides aminés (ici, les fréquences d'apparition dans nos alignements de départ).
On en déduit la vraisemblance d'un alignement entre AAi et AAj : 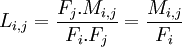
En pratique, on utilise les logarithmes car 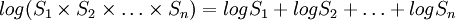 .
.
La matrice de substitution se calcule donc à l'aide de la formule suivante : 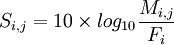
Pour les fréquences de mutations de MM120 correspondant à 120 unités MM1, il faut multiplier 120 fois MM1 avec elle-même (produit vectoriel).
round(MM120*100) A R N D C Q E G H I L K M F P S T W Y V A 32 5 6 6 10 6 7 8 4 5 5 6 5 3 8 11 8 3 3 7 R 3 34 4 3 3 7 4 3 5 2 2 10 3 2 3 4 4 2 2 2 N 3 3 26 6 2 4 4 4 5 1 1 4 2 1 2 4 4 1 2 1 D 4 4 9 34 2 6 11 4 5 1 1 5 2 1 3 5 4 1 2 2 C 1 1 1 0 31 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 Q 3 5 3 4 2 22 5 2 4 1 2 5 3 1 2 3 3 1 2 2 E 6 6 7 13 3 10 31 4 5 2 2 8 3 2 5 6 5 2 3 3 G 7 4 7 5 4 4 5 55 4 1 2 4 2 2 3 7 4 2 2 2 H 1 2 3 2 1 2 2 1 29 1 1 2 1 2 1 1 1 2 4 1 I 4 2 2 2 5 3 2 1 2 32 12 3 10 6 2 3 5 3 4 16 L 5 4 3 2 5 5 3 2 4 15 39 4 18 12 3 4 5 5 7 11 K 5 13 7 6 3 9 8 4 6 3 3 29 4 2 4 6 5 2 3 3 M 1 1 1 1 2 2 1 1 1 4 5 1 22 3 1 1 2 1 2 3 F 2 1 1 1 3 1 1 1 4 4 5 1 5 37 1 1 2 10 14 3 P 4 2 2 2 2 3 3 2 3 1 2 3 1 1 48 4 2 1 1 2 S 7 4 6 5 6 5 5 5 4 2 3 5 3 2 5 25 8 3 2 3 T 5 4 5 4 6 5 4 3 4 4 3 4 4 2 3 8 29 2 3 5 W 0 0 0 0 1 0 0 0 1 0 1 0 1 2 0 0 0 45 2 0 Y 1 1 2 1 2 2 1 1 5 2 2 1 2 10 1 1 1 8 37 1 V 6 3 3 2 8 4 3 2 3 18 10 3 9 6 3 4 7 4 4 32
Ensuite, il n'y a plus qu'à appliquer la formule 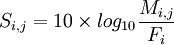 .
.
Vous devriez obtenir la matrice de substitution suivante :
round(S120) A R N D C Q E G H I L K M F P S T W Y V A 6 -2 -2 -2 1 -1 -1 0 -3 -2 -3 -2 -2 -4 0 1 0 -5 -4 -1 R -2 8 -1 -2 -3 1 -1 -3 0 -4 -4 3 -3 -5 -3 -1 -2 -4 -3 -4 N -1 0 9 2 -2 0 0 0 1 -4 -4 1 -3 -4 -2 1 0 -4 -2 -4 D -2 -2 2 8 -4 0 3 -2 -1 -6 -6 -1 -5 -6 -2 0 -2 -6 -4 -6 C 0 -3 -2 -4 14 -4 -4 -3 -3 -1 -2 -4 -2 -1 -4 0 0 0 -1 0 Q -1 2 0 0 -3 8 2 -3 1 -3 -2 2 -1 -4 -1 0 -1 -3 -2 -3 E -1 0 0 3 -4 2 7 -2 -1 -5 -5 1 -4 -6 -2 -1 -1 -5 -4 -4 G -1 -4 -1 -2 -4 -4 -3 8 -4 -8 -7 -3 -7 -8 -4 -1 -4 -6 -7 -7 H -3 0 1 0 -3 1 -1 -3 12 -4 -3 0 -2 0 -2 -1 -2 1 3 -4 I -2 -4 -4 -6 -1 -4 -5 -7 -4 7 2 -4 2 0 -5 -4 -1 -3 -2 4 L -3 -4 -5 -6 -2 -3 -5 -7 -3 2 6 -4 3 1 -4 -4 -3 -2 -1 1 K -1 3 0 0 -3 2 1 -2 0 -4 -3 7 -2 -5 -2 0 -1 -5 -4 -4 M -2 -2 -3 -5 -1 -1 -3 -6 -2 2 3 -2 10 1 -4 -2 -1 -2 -1 1 F -4 -5 -5 -6 -1 -4 -6 -7 0 0 1 -5 1 10 -6 -4 -4 4 6 -2 P -1 -3 -3 -3 -4 -2 -2 -4 -2 -6 -5 -3 -5 -6 10 -1 -3 -7 -6 -4 S 1 -1 1 0 1 0 0 0 -1 -3 -3 0 -2 -4 0 7 2 -3 -3 -2 T 0 -2 0 -1 0 -1 -1 -3 -2 -1 -2 -1 -1 -4 -2 2 7 -5 -3 0 W -6 -4 -5 -6 -1 -4 -6 -6 0 -3 -3 -6 -3 4 -7 -4 -5 17 4 -4 Y -4 -4 -2 -4 -1 -3 -5 -7 3 -3 -1 -4 -1 6 -6 -4 -4 4 11 -3 V -1 -4 -4 -5 0 -3 -4 -6 -4 4 1 -4 1 -1 -4 -3 0 -3 -3 6
Calcul de la matrice des fréquences de mutation et calcul des fréquences d'apparition des acides aminés à partir d'une matrice PAM
Nous allons partir de la matrice File:PAM250.txt similaire à la matrice de substitution que nous avons calculée précédemment.
Comme vu dans la section précédente, nous savons que 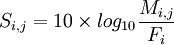 .
.
D'où la différence entre 2 valeurs d'une même ligne de la matrice (PAM250) :
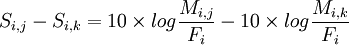
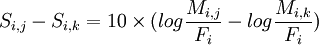
avec 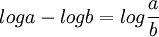
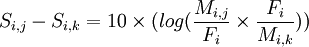
les Fi se simplifient et on obtient :
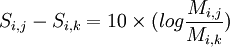
d'où
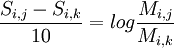
d'où
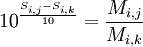
En supposant que Mi,1 = 1 (première colonne de MM à 1), on peut en déduire les autres valeurs de la matrice pour la ligne i de MM. En utilisant la formule ci-dessus avec k = 1 :
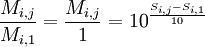
On obtient donc la matrice suivante :
round(MM[1:10,1:10],2) A R N D C Q E G H I A 1 0.44 0.49 0.49 0.68 0.51 0.54 0.59 0.40 0.42 R 1 3.89 1.35 1.12 0.89 1.78 1.38 0.87 1.45 0.65 N 1 1.20 2.63 1.82 0.87 1.29 1.38 1.26 1.48 0.54 D 1 1.00 1.82 3.31 0.63 1.38 2.14 1.00 1.20 0.39 C 1 0.58 0.63 0.46 7.59 0.54 0.49 0.52 0.56 0.81 Q 1 1.51 1.23 1.32 0.71 2.09 1.55 0.74 1.29 0.63 E 1 1.12 1.26 1.95 0.62 1.48 2.19 0.79 1.02 0.46 G 1 0.65 1.05 0.83 0.60 0.65 0.72 4.57 0.59 0.26 H 1 1.58 1.82 1.48 0.95 1.66 1.38 0.87 6.92 0.72 I 1 0.68 0.63 0.46 1.32 0.78 0.59 0.37 0.69 3.31
Il s'agit maintenant d'ajuster ces fréquences pour que leur somme sur une colonne soit égale à 1.
S'il on transpose cette matrice (A = MT), cela correspond à un système linéaire de 20 équations à 20 inconnues : Ax = b avec bi = 1 pour ![i \in [1, 20]](/site/images/math/c/2/c/c2cc8dcde1df82e583781495f8607349.png) :
:
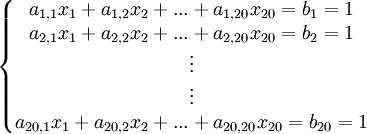
que l'on représente sous forme matricielle : Ax = b
avec
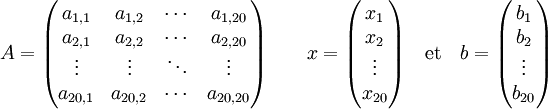
Si A est une matrice carrée inversible, on a un système de Cramer que l'on peut résoudre de la manière suivante :
Ax = b
En multipliant des deux côtés par A − 1, on obtient :
A − 1Ax = A − 1b
or A − 1A = I d'où A − 1Ax = Ix = x = A − 1b
Et nous avons la solution x du système linéaire pour que la somme des lignes de A soit égale à 1 (donc la somme des colonnes de MM soit égale à 1).
Comme A correspond au système suivant :
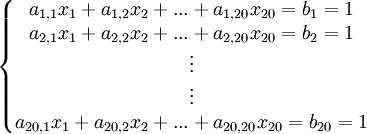
On remarque que cela correspond à multiplier la 1ère colonne de A par x1, la 2ème colonne de A par x2, ...
Et comme A = MT, cela correspond à multiplier la 1ère ligne de MM par x1, la 2ème ligne de MM par x2, ...
Après avoir vérifié que A est inversible, déterminez la matrice MM. Vous devriez obtenir :
round(MM[1:10,1:10],2) A R N D C Q E G H I A 0.15 0.07 0.08 0.08 0.10 0.08 0.08 0.09 0.06 0.06 R 0.04 0.17 0.06 0.05 0.04 0.08 0.06 0.04 0.06 0.03 N 0.04 0.05 0.10 0.07 0.03 0.05 0.05 0.05 0.06 0.02 D 0.05 0.05 0.09 0.16 0.03 0.07 0.11 0.05 0.06 0.02 C 0.02 0.01 0.01 0.01 0.12 0.01 0.01 0.01 0.01 0.01 Q 0.03 0.05 0.04 0.04 0.02 0.07 0.05 0.02 0.04 0.02 E 0.07 0.08 0.09 0.14 0.04 0.11 0.16 0.06 0.07 0.03 G 0.09 0.06 0.09 0.07 0.05 0.06 0.06 0.39 0.05 0.02 H 0.02 0.02 0.03 0.02 0.01 0.03 0.02 0.01 0.10 0.01 I 0.05 0.03 0.03 0.02 0.07 0.04 0.03 0.02 0.04 0.17
Calculons à présent la fréquence d'apparition des a.a. dans une séquence. On sait que
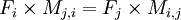
d'où
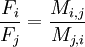
Comme précédemment, on suppose que pour j = 1,Fj = F1 = 1, ce qui nous permet de calculer les autres fréquences : 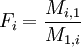
Sachant que la somme des fréquences des a.a. doit être égale à 1, vous pouvez maintenant mettre à l'échelle ces valeurs que l'on vient d'obtenir pour que leur somme soit 1.
Vous devriez obtenir les fréquences suivantes :
round(Freq,2) A R N D C Q E G H I L K M F P S T W Y V 0.08 0.05 0.04 0.05 0.01 0.03 0.07 0.08 0.02 0.07 0.09 0.06 0.03 0.04 0.04 0.05 0.06 0.01 0.03 0.08
Ce qui correspond bien aux fréquences d'a.a. calculées dans la première partie à partir de la matrice CM.



