L2-L3 Bioinfo - TD Bases de donnees - correction
From silico.biotoul.fr
Contents |
Contexte
- sensisibilté/résistances d'écotypes différents d'Araidopsis thaliana (arabette) à une bactérie pathogène.
- photos et mesures de la surface foliaires de différents écotypes
- par la suite, on va chercher à analyser la croissance des plantes, et donc leur sensibilité, par rapport aux génotypes
- stockage de tout ça dans une base de données pour leur analyse statistique par la suite
- en plus de la surface foliaire, il faut donc aussi stocker des informations sur les différents écotypes, les mesures effectuées et des informations sur les génotypes
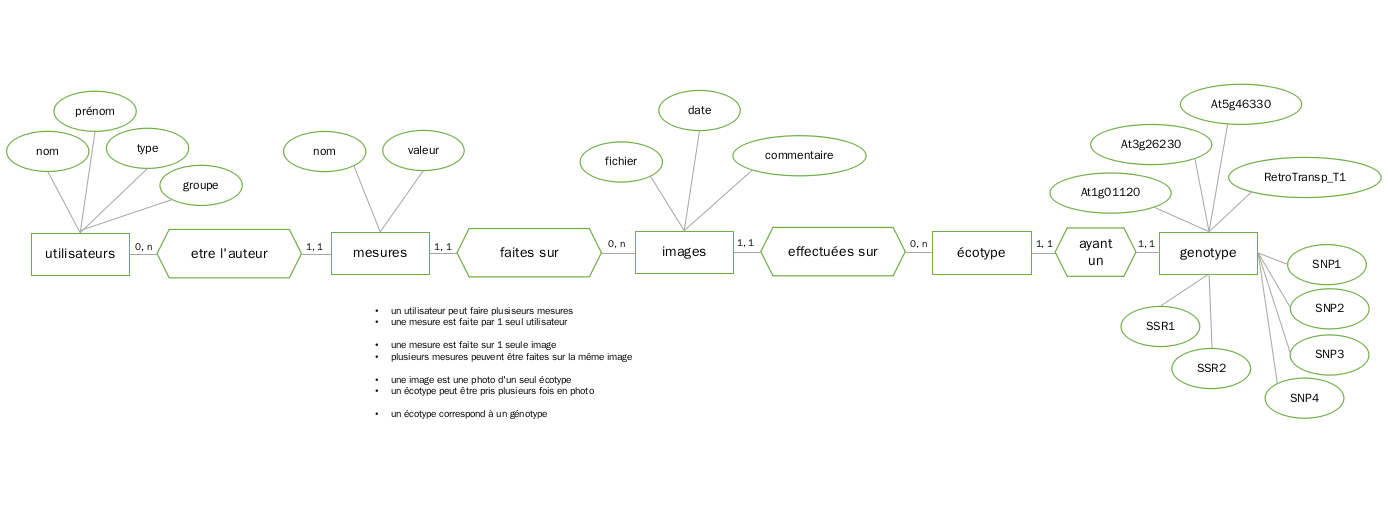
Au niveaux des écotypes, afin de les différencier, à chaque écotype est attribué un identifiant unique (Accession_id) permettant de renseigner le pays d'origine (Country) avec la Latitude et la Longitude exactes ainsi que si la bactérie pathogène est naturellement présente (Natural_Bacterial_occurence). Concernant les images analysées à la 1ère séance de TP, il faut renseigner à quel écotype chaque image correspond, quand a été prise l'image et éventuellement pouvoir ajouter des commentaires. Ensuite, avec les analyses effectuées, on souhaite également enregistrer la surface foliaire mesurée, et par qui. Il faudra donc aussi conserver des informations sur les utilisateurs telles que leur identifiant, leur nom et leur prénom, leur provenance (L2/L3 BCP/2B2M/BOPE) et leur groupe de TP. Une autre mesure déjà effectuée sera stocker aussi : le nombre de racines latérales. Aux niveau des génotypes, on considère un certain nombre de marqueurs :
- présence ou absence des gènes ou loci At1g01120, At3g26230, At5g46330 et RetroTranp_T1 dans le génome,
- la valeur des marqueurs de type SNP (Single Nucleotide Polymophism) pouvant être G, A , T ou C : SNP1 à SNP4
- le nombre de répétitions de marqueurs de type SSR (Simple Sequence Repeat) : SSR1 et SSR2
Modèle entités-associations
Dans un premier temps, proposez un modèle entités-associations modélisant les données à gérées.
Pour cela, il vous faudra identifier les entités (ex: écotype) et leurs attributs. Et en parallèle, les relations/associations entre ces entités avec leurs attributs et la cardinalité des associations (ex: plusieurs images peuvent correspondre au même écotype).
Modèle relationnel
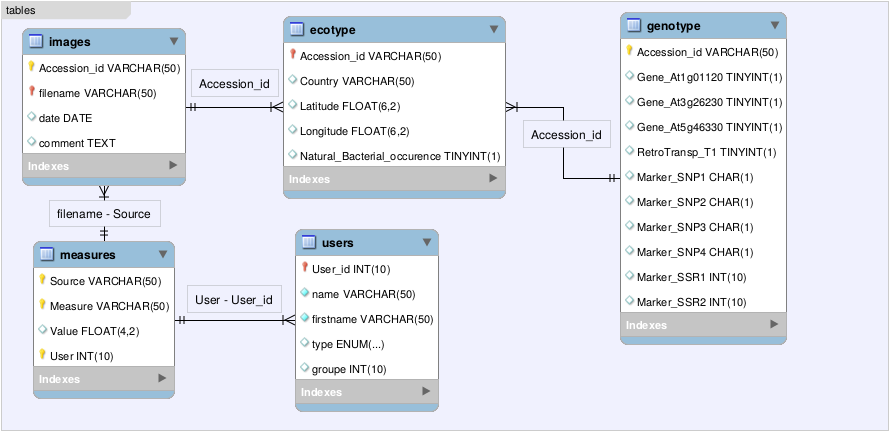
A partir du modèle précédent, il s'agit maintenant de le traduire en modèle relationnel ou schéma de bases de données.
Il faudra donc définir les différentes tables avec leurs colonnes et le type de chaque colonne, ainsi que les attributs constituants les clés primaires (valeur unique dans une table) et les clés étrangères (attributs référençant d'autres lignes dans une table).
Utilisation de la base de données
Consultation
Sélection (des lignes et/ou des colonnes), tri, regroupement et sélection après regroupement :
- Afficher tous les utilisateurs.
SELECT * FROM users;
- Afficher tous les utilisateurs la même provenance que vous (ex: L2 BCP).
SELECT * FROM users WHERE type='TEACHER';
- Afficher tous les identifiants, noms et prénoms des utilisateurs de la même provenance que vous.
SELECT User_id, firstname, name FROM users WHERE type='TEACHER';
- La même chose mais en triant les résultats par nom.
SELECT User_id, firstname, name FROM users WHERE type='TEACHER' ORDER BY name;
- Afficher tous les utilisateurs provenant de L2 triés par nom.
SELECT User_id, firstname, name, type FROM users WHERE type LIKE 'L2%' ORDER BY name;
- La même chose mais triés par provenance puis par nom.
SELECT User_id, firstname, name, type FROM users WHERE type LIKE 'L2%' ORDER BY type, name;
- Afficher les surfaces foliaires mesurées sur les images triés par surface décroissante.
SELECT * FROM measures WHERE Measure='Leaf_area';
- Quels sont le minimum, la moyenne et le maximum de surface foliaire ?
SELECT min(Value), avg(Value), max(Value) FROM measures WHERE Measure='Leaf_area';
- Combien y a-t-il d'utilisateurs dans la table ?
SELECT COUNT(*) FROM users;
- Combien y a-t-il d'utilisateurs par provenance ?
SELECT type, COUNT(*) FROM users GROUP BY type;
- La même chose trié par effectifs décroissant.
SELECT type, COUNT(*) AS effectifs FROM users GROUP BY type ORDER BY effectifs DESC; SELECT type, COUNT(*) AS effectifs FROM users GROUP BY type ORDER BY 2 DESC;
- La même chose ayant au moins 10 utilisateur
SELECT type, COUNT(*) AS effectifs FROM users GROUP BY type HAVING effectifs>9 ORDER BY effectifs DESC;
Pour chez soi :
- combien y a-t-il d'écotypes en tout ou par pays ? puis trié de manière décroissante ; puis, ayant au moins 5 écotypes dans le même pays.
SELECT * FROM ecotype; SELECT Country, COUNT(*) AS nb FROM ecotype GROUP BY Country; SELECT Country, COUNT(*) AS nb FROM ecotype GROUP BY Country ORDER BY 2 DESC; SELECT Country, COUNT(*) AS nb FROM ecotype GROUP BY Country HAVING nb>=5 ORDER BY 2 DESC;
Jointure avec produit cartésien
|
T
|
|
U
|
= |
T
|
Requête SQL pour faire la jointure : filtrer les lignes dont la 1ère colonne de T correspond à la 1ère colonne de U :
SELECT * FROM T, U WHERE Tid = Uid
Résultat :
| Tid | Tcol2 | Uid | Ucol2 |
|---|---|---|---|
| 1 | a | 1 | X |
| 1 | b |
1 | X |
| 2 | c | 2 | Y |
Jointure avec JOIN
Le même résultat est obtenu avec JOIN en précisant les colonnes qui doivent correspondre :
SELECT * FROM T JOIN U ON (Tid=Uid)
Résultat :
| Tid | Tcol2 | Uid | Ucol2 |
|---|---|---|---|
| 1 | a | 1 | X |
| 1 | b |
1 | X |
| 2 | c | 2 | Y |
Questions :
- Afficher les mesures vous avez effectuées.
SELECT * FROM measures m JOIN users u ON (m.User = u.User_id) WHERE u.name='BARRIOT';
- Afficher les mesures effectuées par les personnes du même groupe de TP que vous.
SELECT * FROM measures m JOIN users u ON (m.User = u.User_id) WHERE groupe=0;
- Afficher les mesures concernant des écotypes pour lesquels la bactérie est naturellement présente.
SELECT * FROM measures m JOIN images i ON (m.Source=i.filename) JOIN ecotype e ON (i.Accession_id = e.Accession_id) WHERE Natural_Bacterial_occurence=1;
- De même, mais ayant le marqueur SNP1 valant A
SELECT * FROM measures m JOIN images i ON (m.Source=i.filename) JOIN ecotype e ON (i.Accession_id = e.Accession_id) JOIN genotype g ON (e.Accession_id=g.Accession_id) WHERE Natural_Bacterial_occurence=1 AND Marker_SNP1='A';
Synthèse des éléments d'une requête
SELECT -- choix des colonnes. ex: Accession_id, Country FROM -- choix des tables. ex: ecotype WHERE -- filtre éventuel sur les lignes à afficher ex: Bacterial_Natural_occurence = 1 GROUP BY -- regroupement éventuel de lignes ex: Accession_id HAVING -- filtre sur les valeurs des lignes regroupées ex: COUNT(*)>1 ORDER BY -- tri des résultats ex: Country DESC
Ajout de données
Exemple de requête :
INSERT INTO ecotype ('Accession_id', 'Country') VALUES ('patient_0', 'France');
Mise à jour
Exemple de requête :
UPDATE ecotype SET Bacterial_Natural_occurence=1 WHERE Accession_id='patient_0';