L2-L3 Bioinfo - TP Bases de donnees
From silico.biotoul.fr
Contents |
Création de la base de données
Afin de créer une base de données, connectez vous sur le site http://localhost/phpmyadmin/ ou http://localhost/phpMyAdmin
Problème depuis 2021-22 : ce n'est pas installé sur les machines en TP. Il ne sera donc pas possible de travailler sur le serveur Web et le serveur de bases de données de votre PC en salle TP.
Vous allez donc travailler sur le serveur commun à tous les étudiants dont le lien se trouve sur la page http://silico.biotoul.fr/enseignement/lbioinfo/data-links.php. A partir de phpMyAdmin, créez une base de données ayant pour nom votre login moodle (par exemple BRR4409A) pour faire la 1ère partie du TP (Création d'une base, d'une table, sauvegarde et restauration).
Important : Pour cette partie où normalement chacun·e est sur son serveur tout·e seul·e, n'allez pas abîmer les bases de données de vos collègues ni celle qui servira pour la 2ème partie (nommée lbioinfo) et qui est destinée à recueillir toutes les mesures de tout le monde pour ensuite s’entraîner sur les requêtes de consultations.
Il s'agit d'un serveur Web local (installé sur votre ordinateur). Le programme faisant office de serveur Web s'appelle Apache. Des pages Web dynamiques fournissent une interface Web de gestion de bases de données (phpMyAdmin) pour un serveur de bases de données MySQL ou MariaDb (l'équivalent open source).
Ces programmes sont open sources et/ou gratuits et peuvent être installés sur Windows, Linux ou MacOS. Il existe des projets les rassemblant et qui permettent donc une installation rapide pour les néophytes : http://www.wampserver.com pour Windows par exemple.
Pour commencer, il faut créer une nouvelle base de données. Pour cela, cliquez sur l'onglet Bases de données. Vous avez alors la possibilité de nommer cette nouvelle base et de cliquez sur le bouton Créer. Cette nouvelle base devrait ensuite s'afficher sur la gauche de la page avec les autres bases existantes (information_schema, mysql et test normalement).
Vous pouvez alors cliquer sur cette nouvelle base pour la sélectionner. Il devrait s'afficher une page vous proposant de créer une nouvelle table.
Si vous désirez supprimer une base, vous pouvez après l'avoir sélectionnée, aller dans l'onglet Opérations, et cliquez sur Supprimer la base de données.
Création de tables
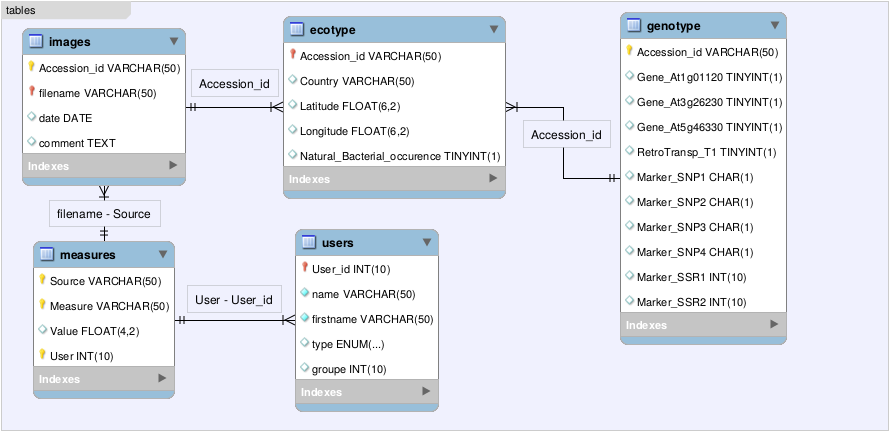
Pour cela, il faut sélectionner une base de données. Puis, dans l'onglet Structure, indiquez le nom de la table ainsi que le nombre de colonnes. D'où l'importance de déterminer à l'avance le schéma de la base de données. Néanmoins, la plupart des systèmes de gestion de bases de données (SGBD) actuels autorisent la modification de la structure de la base (ajout, suppression, renommage de colonnes et autres).
Créez la table ecotype définit dans le schéma ci-dessous.
Sauvegarde et restauration d'une base de données
La sauvegarde (appelée souvent dump) se fait dans l'onglet Exporter. Selon que vous êtes sur la page de la base ou sur la page d'une table, l'export se fera soit de la base complète soit de la table sélectionnée.
L'export permet de sauvegarder une image de la structure et des données de la base, ce qui permet de la restaurer sur un autre serveur de base de données (ou le même).
Essayez donc en cliquant sur l'onglet Exporter avec les options par défaut. Enregistrez puis/ou visualisez le fichier avec un éditeur de texte. Il s'agit du langage SQL et des commandes permettant la création de(s) table(s) (et éventuellement l'insertion des données si elles en contiennent). Les principales commandes sont donc :
- CREATE TABLE nom_de_la_table (nom_colonne type_colonne, ...
- INSERT INTO nom_de_la_table (noms des colonnes concernées) VALUES (valeurs pour ces colonnes)
Pour cette partie du TP, vous allez travailler sur une sauvegarde contenant à la fois la structure (création des tables) et les données que vous compléterez plus tard avec vos mesures (sur le serveur partagé par tout le monde).
Vous allez donc restaurer cette image en cliquant sur l'onglet Importer et en fournissant le fichier correspondant à la sauvegarde.
Après avoir restauré cette image, allez sur la page de la base lbioinfo. Combien y a-t-il de lignes dans chacune des table ?
Explorez les données et essayez de déterminer quel écotype est le moins sensible.
Edition manuelle des données
Vous allez à présent ajouter les mesures de surface foliaire que vous avez effectuées sur les images au TP précédent.
Les liens vers les mesures saisies devraient se trouver encore sur la page http://silico.biotoul.fr/enseignement/lbioinfo/data-links.php
Sur cette page, vous y trouverez également un lien vers l'interface de phpMyAdmin sur le serveur silico. Connectez-vous (même identifiant et mot de passe que accéder à la page des liens), puis utiliser la base de données lbioinfo. Sélectionnez la table measures. C'est dans celle-ci que vous allez ajouter vos mesures de surface foliaire.
Une fois la table sélectionnée, allez dans l'onglet Browse. Notez que
- la colonne Source va correspondre aux noms des fichiers que vous avez analysés
- la colonne Measure devra avoir la valeur Leaf_area en respectant bien les majuscules et minuscules
- la colonne Value prendra la valeur de la surface foliaire que vous avez mesurée en cm2
- la colonne User devra contenir votre identifiant. ATTENTION, cet identifiant est différent de votre numéro étudiant, il faut aller le chercher dans la table users.
Pour insérer vos mesures, allez dans l'onglet Insert.
Requêtes de consultation
Après avoir ajouté vos mesures, commencez par essayer les requêtes SQL élaborée pendant la séance du TD précédent (onglet SQL):
- Afficher tous les utilisateurs
- Afficher tous les utilisateurs la même provenance que vous (ex: L2 BCP)
- Afficher tous les identifiants, noms et prénoms des utilisateurs de la même provenance que vous
- La même chose mais en triant les résultats par nom.
- Afficher tous les utilisateurs provenant de L2 triés par nom.
- La même chose mais triés par provenance puis par nom.
- Afficher les surfaces foliaires mesurées sur les images triés par surface décroissante.
- Quels sont le minimum, la moyenne et le maximum de surface foliaire ?
- Combien y a-t-il d'utilisateurs dans la table ?
- Combien y a-t-il d'utilisateurs par provenance ?
- La même chose trié par effectifs décroissant.
- La même chose ayant au moins 10 utilisateur
- Afficher les mesures que vous avez effectuées.
- Afficher les mesures effectuées par les personnes du même groupe de TP que vous.
- Afficher les mesures concernant des écotypes pour lesquels la bactérie est naturellement présente.
- De même, mais ayant le marqueur SNP1 valant A
- Afficher les mesures des écotypes où la présence d'un de ces deux gènes (At1g01120, At3g26230 ) est avéré
- Afficher la variance et la moyenne des surfaces foliaires par écotype
- Afficher le nombre de mesures par écotype
- Afficher les noms des étudiants qui ont enregistré les mesures d'un écotype en particulier, par exemple Ita-0
Rappel sur la syntaxe d'une requête de consultation SQL
SELECT -- choix des colonnes. ex: Accession_id, Country FROM -- choix des tables. ex: ecotype WHERE -- filtre éventuel sur les lignes à afficher ex: Bacterial_Natural_occurence = 1 GROUP BY -- regroupement éventuel de lignes ex: Accession_id HAVING -- filtre sur les valeurs des lignes regroupées ex: COUNT(*)>1 ORDER BY -- tri des résultats ex: Country DESC
Pour une opération de jointure :
SELECT * FROM tab_1 JOIN tab_2 ON tab_1.col_A = tab_2.col_B
Pour les opérations de regroupement :
SELECT id, COUNT(*) AS effectifs, MAX(colonne), MIN(colonne), AVG(colonne), STDDEV(colonne) FROM tab GROUP BY id
Note : AVG pour la moyenne et STDDEV pour l'écart-type.
Recherche de corrélation entre le phénotype et le génotype
Pour la suite des analyses, nous allons nous intéresser à la recherche de causes génotypiques pouvant expliquer les phénotypes observer.
Les informations traitées seront analysées à partir de tableaux de données extraits de la base de données et des mesures que vous avez réalisées. Un tableau synthétique peut être obtenu avec la requête suivante :
SELECT e.Accession_id AS Accession_id,e. Latitude AS Latitude, e.Longitude AS Longitude, m1.Value AS Lateral_Root_number, avg(m2.Value) AS Leaf_area, e.Natural_Bacterial_occurence AS Natural_Bacterial_occurence, g.Gene_At1g01120 AS Gene_At1g01120, g.Gene_At3g26230 AS Gene_At3g26230, g.Gene_At5g46330 AS Gene_At5g46330, g.RetroTransp_T1 AS RetroTransp_T1, g.Marker_SNP1 AS Marker_SNP1, g.Marker_SNP2 AS Marker_SNP2, g.Marker_SNP3 AS Marker_SNP3, g.Marker_SNP4 AS Marker_SNP4, g.Marker_SSR1 AS Marker_SSR1, g.Marker_SSR2 AS Marker_SSR2 FROM ecotype e JOIN images i1 ON (e.Accession_id = i1.Accession_id) JOIN measures m1 ON (i1.filename = m1.Source) JOIN users u1 ON (m1.User = u1.User_id) JOIN images i2 ON (e.Accession_id = i2.Accession_id) JOIN measures m2 ON (i2.filename = m2.Source) JOIN users u2 ON (m2.User = u2.User_id) JOIN genotype g ON(e.Accession_id = g.Accession_id) WHERE m1.Measure = 'Lateral_Root_number' AND m2.Measure = 'Leaf_area' GROUP BY e.Accession_id;
Comme le résultat d'un requête correspond aussi a une table, il est possible de "sauvegarder" ces requêtes que l'on appelle "vue". Le tableau précédent est obtenu à partir de la vue "overview" de la base de données que vous avez manipulé.