M1 MABS BBS Data Mining Clustering
From silico.biotoul.fr
Contents |
Travail à effectuer
- Effectuer un clustering sur le jeu de données Heart (avec Knime ou autre)
- Calculer le coefficient de silhouette (par individu, par cluster, et pour le partitionnement)
- Calculer l'entropie et la pureté
- Comparer le partitionnement avec les classes prédéfinies avec un coefficient simple d'appariement et avec l'indice de Jaccard
Silhouette coefficient
- for i-th object, calculate its average distance to all other objects in its cluster. Call this value ai
- for i-th object and any cluster not containing the object, calculate the object's average distance to all the objects in the given cluster. Find the minimum such value with respect to all clusters. Call this value bi
- for the i-th object, the silhouette coefficient is si = (bi - ai) / max(ai, bi)
The value can vary between -1 and 1. Negative values are bad because it means that the average distance to the points in the cluster is greater than the average distance to the points in another cluster. Thus, we want the silhouette coefficient to be positive and ai to be as close to 0 as possible since the coefficient assumes its maximum value of 1 when ai = 0.
- Silhouette coefficient for a cluster
Simply average the silhouette coefficient of all the points in that cluster.
- goodness of a clustering
Simply average the silhouette coefficient of all the points.
Entropie
pij = mij / mi probabilité qu'un membre du cluster i appartienne à la classe j, avec mi la taille du cluster i, et mij le nombre d'objets de la classe j dans le cluster i.
Entropie du cluster i: 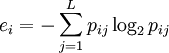 avec L le nombre de classes
avec L le nombre de classes
Entropie totale: 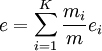 avec K le nombre de clusters
avec K le nombre de clusters
Pureté
pij comme pour l'entropie.
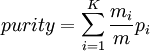 avec pi = maxjpij et K le nombre de clusters
avec pi = maxjpij et K le nombre de clusters



