M2R ADAM
From silico.biotoul.fr
Introduction
Le but de ce TD est de se familiariser avec les concepts et les outils de biologie moléculaire nécessaires à l’analyse de séquences nucléiques (clonage, analyse de restriction, alignement...) par utilisation de logiciel dédié.
La version gratuite académique de BenchLing sera utilisé pour ce TD. D'autres logiciels comme Serial Cloner , CLC, / pDRAW32 sont également disponibles pour réaliser ce type d'analyse.
Installation de BenchLing et récupération des données
- Connectez vous à BenchLing, et créez vous un compte sur la version 'Académique' (For Academia > Sign In).
- Ouvrez BenchLing, et récupérer les données necessaires pour le TD
Solliciter une participation à l'organisation LRSVEnseignement
Dans benchling, en bas a gauche, cliquez sur le rond contenant votre initiale de compte Puis sur le signe +, et Join Organization Rechercher le terme LRSV Sélectionner LRSVEnseignement Attendez confirmation par l'enseignant de votre acceptation a l'orgnisation LRSVEnseignement
Récupérer les données (non modifiables) pour le TD et créer un nouveau répertoire de données (modifiables)
Dans benchling, en haut a gauche, cliquez sur le logo 'valise' Vous devez voir apparaitre une valise LRSVEnseignement Shared project et d'autres anciens répertoires ! Cliquer sur la valise correspondante; vous devez voir un ensemble de fichier apparaitre (IPM60, IPM61...) Nous allons maintenant créer un nouveau répertoire qui comprendra l'ensemble de ces fichiers pour pouvoir les manipuler Cliquer sur le signe + (colonne bleue a gauche) > 'Project' Identifiez votre projet personnel, nommer le par exemple 'Nom Prenom '
Donner les droits sur vos fichiers
Cliquer sur 'all project', vous devez avoir maintenant 2 projets LRSVEnseignement Shared project & Nom Prenom Paramétrer les droits sur ce répertoire (NB :tout doit etre administre par LRSVEnseignement, pour que l'enseignant puisse supprimer les fichiers en fin d'année !), en cliquant bouton droit de la souris 'Settings' Owner = LRSVEnseignement / Members of LRSV Enseignement et tous les autres = ADMIN
Copier des fichiers dans votre répertoire
Copier l'ensemble des fichiers de LRSVEnseignement dans votre répertoire personnel Nom Prenom:
Cliquer sur le logo copie (en haut a droite) > sélectionnez les fichiers > copy vers votre répertoire personnel
OU
Sélectionner tous les fichiers depuis le repertoire LRSVEnseignement > en haut a droite petit carré, 'copy to..' vers votre répertoire
Revener dans votre répertoire personnel; vous devez avoir maintenant les 9 fichiers sur lesquels vous allez travailler
pendant le TD (Copy of IPM60, Copy of...)
Prise en main de BenchLing
- Regardez les principales fonctions du logiciel en travaillant avec la carte du plasmide pUC19
Editer une carte de restriction et les annotations
Aller dans votre réperoire personnel (logo valise)
Cliquer sur le fichier pUC19 : l'écran est séparé en 2 parties (gauche = séquence, droite = carte du plasmide).
Si ce n'est pas le cas cliquer sur SplitWorkspace (en bas droite ecran)
Partie Droite (Carte) : Tester les fonctions de l'onglet du haut (linear map, plasmid, description..)
Tester l'onglet juste en dessous (roue/paramètres) et faites afficher les différents paramètres de la carte du plasmide (ORF, site de restiction...)
Sur la carte, cliquez sur le marqueur de selection AmpR (Ampicilline)
Notez que la séquence est surlignée sur la partie gauche de l'écran (zone modifiable: etendre/restreindre avec la souris clic)
Vice versa, sur la séquence, cliquez sur le gène de sélection LacZ. Modifier la zone sélectionnée
L'outil colonne de droite, partie droite de l'écran : les enzymes de restriction
Editer les enzymes de restriction du vecteur en cliquant sur le logo 'ciseau' Réalisez une digestion virtuelle de pUC19 avec l'enzyme commercialisée par NEB, nommée AclI.
- Combien de site de restrictions ?
- Taille des Fragments générés?
- Position sur le vecteur?
- Visualisation du gel d'électrophorèse
Réalisez la double digestion AclI & BamH1.
- Les tampons sont-ils compatibles ?
- Quelle est la température optimale de digestion?
- Nombre de fragments et taille des fragments obtenus?
L'outil en bas a droite de l'écran, partie droite de l'écran : clonage/assembly wizard
Nous allons cloner/liguer un insert issu du vecteur pATS-CBEL (carte disponible) dans le vecteur de destination pUC19 (carte disponible)
Une double digestion par les enzymes de restriction BamH1 + HindIII (site de coupure unique) est réalisée (=clonage orienté)
Ouvrer les 2 cartes (pUC19 et pATS-CBEL). Notez les 2 onglets en haut de page
En restant sur la carte de pUC19, cliquez sur 'Assembly Wizard'> Create a New Assembly > Digest and Ligate (Notez que vous pouvez faire du Golden Gate)
En bas de l'écran s'ouvre 3 onglets : SET FRAGMENT = position de la zone sélectionnée pour le clonage
INSERT = vecteur d'où provient l'insert a cloner (pATS-CBEL)
BACKBONE = vecteur de destination (pUC19)
Specifier le BACKBONE (pUC19): sélectionnez la région du vecteur pUC19 qui va être liguée avec l'insert et se situant entre les sites de restriction Hind III / BamH1 (environ 2,7 kb) (shift clic sur la région)
Spécifier l'INSERT (depuis pATS-CBEL): sélectionnez dans le vecteur pATS-CBEL, la zone correspondant à l'insert et située entre les sites de restriction BamH1/ Hind III (357 pb (shift clic) NB: une autre possibilité pour sélectionner vos fragments est de réaliser la digestion virtuelle et de cliquer sur les régions que vous souhaitez liguer (BACKBONE et INSERT)
Cliquer sur Assemble (petit logo vert en bas a droite) Visualiser la nouvelle carte du plasmide recombinant généré Nommez votre nouveau plasmide pUC19_CBEL (bouton 'i' colonne de droite, droite écran).
- Quelle est la taille du plasmide recombinant pUC19_CBEL?(3013 bp)
- Retrouvez vous les sites de restriction HindIII et BamH1 ?
Contexte Scientifique du TD
Une étude biochimique a permis d’isoler une protéine pariétale de l’oomycète Phytophthora parasitica, parasite racinaire de nombreuses solanacées. Cette protéine, nommée CBEL (Celluose-Binding Elicitor Lectin), est reconnue par la plante (=effecteur), entraînant une mort localisée de certaines cellules végétales. Pour poursuivre les études une protéine recombinante doit être produite. On se propose ici de cloner l'ADNc de CBEL dans le vecteur d’expression pVX (virus X de la pomme de terre) pour obtenir le vecteur pVX-CBEL.
Différentes étapes seront réalisées pour mener à bien ce projet.
Etape 1 : choix des enzymes de restriction adaptées pour le clonage dans le vecteur pVX Etape 2 : création d'une carte du plasmide pVX-CBEL Etape 3 : identification in silico des clones recombinants positifs Etape 4 : définition d'amorce Etape 5 : séquençage Etape 6 : analyse des résultats
Etape 1: choix des enzymes de restriction adaptées pour le clonage dans le vecteur pVX
- L'ADNc pleine taille de CBEL est présent dans un vecteur nommé pBlueScript_SK (pBSK, 3 736pb), présentant un gène codant une résistance à l’ampicilline.
- Le carte du vecteur pVX d'expression, et son site multiple de clonage (MCS), ainsi que la séquence partielle (MCS + insert CBEL) du plasmide pBSK_CBEL sont disponibles ici.
Q1
- Décrire les principales étapes du clonage à réaliser.
- Indiquez comment vous sélectionnez les clones recombinants.
- Afin d’identifier les sites de restriction compatible entre le vecteur d'entrée pBSK_CBEL comportant l'insert CBEL et le vecteur de destination pVX, nous allons visualiser les différents site de restriction présents via BenchLing.
Créer la carte de la zone du vecteur pBlueScript_SK contenant l'insert CBEL dans BenchLing
Placez vous dans votre répertoire personnel, cliquez sur le logo 'valise', colonne de gauche
Cliquez sur le signe + (colonne de gauche)> DNA sequence > New DNA Sequence > Entrez le nom de votre fragment plasmidique : pBSK_CBEL > Linear (et non circulaire)
Dans topology, selectionnez 'linear' > Copier coller votre séquence pBSK_CBEL (partie gauche de la zone de travail)
Identifiez les sites de restriction à coupure unique sur pBSK_CBEL
Cliquez sur le logo 'ciseau' (colonne de droite)
Identifiez les enzymes présentant un site de coupure unique, et disponible chez le fournisseur NEB
Q2
- Quels sont les sites de restriction utilisables avec des enzymes de restriction de Proméga, utilisables pour le clonage de l'insert CBEL dans le vecteur pVX ?
Etape 2: création d'une carte du plasmide pVX-CBEL
L'identification des clones recombinants contenant l'insert CBEL peut s'effectuer à la paillasse par une digestion enzymatique des clones candidats pVX_CBEL ou encore par PCR.
Pour prédire la taille des inserts libérés par analyse de restriction, une digestion in silico du vecteur recombinant pVX_CBEL peut-réalisée. Il faut donc créer la carte du nouveau plasmide.
Utilisation de BenchLing pour créer des cartes de plasmide apres clonage in silico
Ouvrez vos deux fichiers: le vecteur pVX (vecteur circulaire) et la zone du site multiple de clonage contenant l'insert CBEL (pBSK_MCS_CBEL)
Créer la carte du vecteur recombinant qui se nommera pVX_CBEL_M2 en utilisant l'outil de clonage 'Assemble/ligate'
Nommez votre nouveau vecteur pVX_CBEL_M2 (colonne de droite bouton 'i') > Name
- Quelle est la taille du plasmide pVX_CBEL_M2 ? (11 917 pb)
NB : En cas de nécessité vous disposez dans votre répertoire du vecteur recombinant a obtenir : fichier 'PVX_CBEL'
Etape 3: identification in silico des clones recombinants positifs
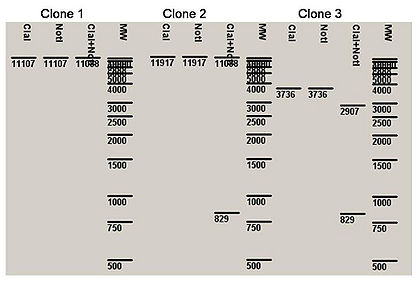
Le clonage et la transformation d’E. coli ont été réalisés avec le vecteur pVX_CBEL. Les colonies bactériennes ont été étalées sur milieu sélectif. L’ADN plasmidique de 3 clones recombinants positifs putatifs a été extrait et digéré via les 2 enzymes de restriction utilisées pour le clonage. Afin de définir le profil de restriction d'un clone recombinant positif, une digestion in silico du vecteur pVX_CBEL peut-être réalisée.
Réaliser une digestion in silico (profil de restriction) du vecteur recombinant pVX_CBEL
Sur le fichier pVX_CBEL, réalisez une digestion avec les 2 enzymes d'intérêt.
Pour cela cliquer sur le logo ciseau (a droite) > sélectionnez vos enzymes > Run Digest
Visualiser le gel d'électrophorèse en cliquant sur l'onglet 'virtuel digest' (juste en dessous du nom du fichier)
- Indiquez, le nombre et les tailles des fragments obtenus après double digestion, ou simple digestion
Ci-dessous le profil de restriction des 3 clones, est présenté :
Q3
- Comment peut-on interpréter ces résultats ?
Q4
- Que faut-il faire pour la suite des expériences ?
Etape 4: définition d'amorces
Pour valider les clones positifs, une vérification par séquençage de la zone d'intérêt est nécessaire. Il faut donc disposer d'amorces (oligonuclétoides) adéquats pour le séquençage. Différents logiciels sont disponibles pour définir des amorces dont PrimerBlastqui présente l'avantage de combiner un logiciel de définition de primer disponible sur le Web (Primer 3), à une analyse de type Blast de vos primers sur les banques du NCBI pour en vérifier leurs spécificités. Vous pouvez aussi définir des oligonucléotides sur Benchling
Utilisation de BenchLing pour l'obtention d'oligonucléotides
Sur le fichier pVX_CBEL_M2 (ou le fichier de correction pVX_CBEL), identifiez les positions de votre insert CBEL
et donc la zone pour définir des oligonucléotides (= de part et d'autre de votre insert CBEL)
Cliquer sur le logo 'double fleche' (colonne de droite) > Create Primers > Wizard > Sequencing (utiliser les paramètres par défaut
OU compléter les paramètres)> Generate primer
Noter que vous pouvez modifier de nombreux paramètres, même la concentration saline de votre réaction
Visualiser les primers/oligonucleotides obtenus sur votre séquence > Cliquez sur le jeu de primers (doite ecran)
Noter l'affichage des primers sur la séquence (gauche de l'écran) sous forme de flèches
Q5
- Définir des oligonucléotides permettant le séquençage de la zone d'intérêt en 5' et 3' (jonction vecteur/insert ; insert CBEL ; jonction insert / vecteur) de pVX_CBEL
Optionnel
Utilisation de PrimerBlast pour l'obtention d'oligonucléotides
Copier-coller la séquence du vecteur pVX_CBEL au format fasta, dans le grand cadre blanc en haut de page
Définir dans "range" les positions de la zone à amplifier
Définir la taille de l'insert à amplifier (sans importance pour le séquençage, indispensable pour une PCR ou qPCR)
Définir le Tm de vos primers (paramètres par défaut)
Sélectionnez les banques à utiliser pour l'analyse Blast (Organism : all, database : nr)
Modification du format de séquences (fasta, texte..)
Utilisez le logiciel SeqRet
Copier-coller la séquence pVX_CBEL
Choisir le format de sortie "pearson/fasta"
Etape 5: séquençage
Un clone dont le profil de digestion ClaI/ NotI est en accord avec la carte de restriction pVX_CBEL, va être séquencé. Les séquençages peuvent s'effectuer sur le site de la plateforme technique Génotoul de Toulouse Midi-Pyrénées ou via d'autres prestataires.
Les résultats de séquençage sont généralement téléchargeables sur votre ordinateur au laboratoire. Avant toute analyse de séquence il est impératif de vérifier la qualité du séquençage (Phred) et de visualiser votre séquence par lecture et interprétation des chromatogrammes. Vous trouverez différents sites internet vous permettant de réaliser ces opérations. Vous trouverez dans ce guide quelques exemples de chromatogrammes, et les soucis souvent rencontrés au séquençage.
Etape 6: analyse des résultats
Avant de continuer les expériences à la paillasse, il sera impératif de BIEN VERIFIER votre clonage et donc que l'insert cloné correspond bien à l'ADNc de CBEL, que l'insert est bien orienté et positionné dans le vecteur pVX entre les sites de restriction ClaI et NotI et DE VISUALISER la qualité de vos chromatogrammes.
Analyse des résultats de séquencage par Blast
Analysez le résultat du séquençage IPM60 (et IPM61) par Blast, sur la banque nr
Pour cela sélectionnez le fragment de séquence que vous souhaitez analyser, cliquer sur 'Blast' (au dessus de la séquence).
Si besoin les séquences IPM60 et IPM61 au format Fasta sont disponibles sur ce lien.
NB: Une autre possibilité est de faire un Blast directement au NCBI : Blast, sur la banque nr
Q6
- Que pensez-vous du résultat Blast ?
- Avec quelle amorce la séquence IPM61 a-t-elle été obtenue ?
Compte-tenu des résultats, il est préférable de réaliser un alignement multiple entre IPM_60 et IPM_61 et le vecteur de destination pVX_CBEL afin de valider les zones de jonction insert/vecteur et le vecteur de destination.
Réalisez un alignement multiple dans BenchLing (Clustal ou MAFT)
Créer un alignement multiple avec le vecteur recombinant PVX_CBEL (vecteur de destination), IPM60 et IPM61
Colonne de droite, cliquez sur le logo 'alignement' > Selectionnez les 3 fichiers requis pour l'alignement multiple
Générer un alignement sous MAFT (reverse/complementation possible) et sous Clustal
Si besoin les séquences IPM60 et IPM61 au format Fasta sont disponibles sur ce lien.
Q7
- Que pouvez vous conclure ?