M2R BV
From silico.biotoul.fr
Introduction
Le but de ce TD est de se familiariser avec les concepts et les outils de biologie moléculaire nécessaires à l’analyse de séquences nucléiques. Ces approches vous seront utiles pendant le M2 ADAM dans le cadre de l’analyse de publications scientifiques et/ou au cours de votre stage. Les exercices s’appuient sur une étude menée actuellement sur le campus Toulousain dans l’UMR5546 et font appel aux moyens disponibles sur le site du Pôle de Biotechnologie Végétale de Toulouse. Ainsi l’ensemble des outils utilisés au cours de ce travail sera accessible depuis les ordinateurs de vos différentes équipes d’accueil du M2 ADAM.
/ pDRAW32 sera utilié comme logiciels pour ce TD, d'autres logiciels (exemple: Serial Cloner , CLC, BenchLing) seront également disponibles dans vos équipes d'accueil.
Contexte Scientifique du TD
Une étude biochimique a permis d’isoler une protéine pariétale de l’oomycète Phytophthora parasitica, parasite racinaire de nombreuses solanacées. Cette protéine, nommée CBEL, est reconnue par la plante (=effecteur), entraînant une mort localisée de certaines cellules végétales. Pour poursuivre les études une protéine recombinante doit etre produite. On se propose ici de cloner l'ADNc de CBEL dans le vecteur d’expression pVX (virus X de la pomme de terre) pour obtenir le vecteur pVX-CBEL.
Différentes étapes seront réalisées pour mener à bien ce projet.
Etape 1 : choix des enzymes de restriction adaptées pour le clonage dans le vecteur pVX Etape 2 : création d'une carte du plasmide pVX-CBEL Etape 3 : identification in silico des clones recombinants positifs Etape 4 : définition d'amorce Etape 5 : séquençage Etape 6 : analyse des résultats
Etape 1: choix des enzymes de restriction adaptées pour le clonage dans le vecteur pVX
- L'ADNc pleine taille de CBEL est présent dans un vecteur nommé pBlueScript_SK (pBSK, 3 736pb), présentant un gène codant une résistance à l’ampicilline.
- Le carte du vecteur pVX d'expression, et son site multiple de clonage (MCS), ainsi que la séquence partielle (MCS + insert CBEL) du plasmide pBSK_CBEL sont disponibles ici.
Q1
- Décrire les principales étapes du clonage à réaliser.
- Indiquez comment vous sélectionnez les clones recombinants.
- Afin d’identifier les sites de restriction compatible entre l’insert CBEL et le vecteur pVX, nous allons visualiser les cartes de restriction des différents plasmides en utilisant le logiciel pDRAW32.
Utilisation de pDRAW Enregistrez sur votre ordi puis ouvrir dans pDRAW le fichier test nommé pATS_CBEL.PDW. NB : les fichiers lisibles dans pDRAW présentent l'extension .PDW Tester les différents fonctions de pDRAW (DNA linear, circular, restriction enzyme...)
Q2
- Via pDRAW, identifiez les sites de restriction compatibles entre le vecteur pBSK_CBEL et du MCS de PVX.
- Sauvegarder pour la suite du TD, la carte du MCS_insert CBEL de pBSK (fichier : MCS_pBSK_CBEL.pdw).
- En cas de nécessité vous pouvez télécharger le fichier de correction MCS_pBSK_CBEL.
Etape 2: création d'une carte du plasmide pVX-CBEL
L'identification des clones recombinants contenant l'insert d'interet CBEL peut s'effectuer à la paillasse par une digestion enzymatique des clones candidats pVX_CBEL. Pour prédire la taille des inserts libérés par digestion, il faut définir la carte du plasmide pVX_CBEL via pDRAW, et ensuite réaliser une digestion 'in silico' avec les enzymes adéquates.
Utilisation de pDRAW pour créer des cartes de plasmide Enregistrez sur votre ordinateur, puis ouvez dans pDRAW carte du plasmide pVX Repérez où se situe le MCS dans pVX Puis dans File, New, Cloning of sequences First Fragment, Use current Open File (soit le vecteur PVX), sélectionner ensuite, 5’ cut : NotI, 3’ cut : ClaI, DNA is circular Cliquez sur Second Fragment, Import Fragment (rechercher votre carte MCS_pBSK_CBEL.pdw) , sélectionner ensuite 5’ cut : ClaI, 3’ cut : NotI, décocher DNA is circular Cliquez sur OK : pour vérifier que votre carte plasmidique est correcte, vous devez retrouver les sites de restriction et CBEL positionné dans le MCS. Changer le nom de votre plasmide par : PVX_CBEL et sauvegarder la carte sous le nom pVX_CBEL En cas de nécessité vous pouvez télécharger le fichier de correction pVX_CBEL.
Etape 3: identification in silico des clones recombinants positifs
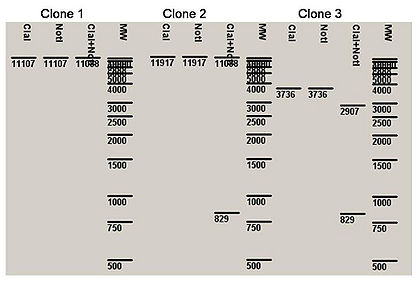
Le clonage et la transformation d’E. coli ont été réalisés. Les colonies bactériennes ont été étalées sur milieu sélectif. L’ADN plasmidique de 5 clones recombinants positifs putatifs a été extrait et digéré via différentes enzymes de restriction. Le profil de digestion de 3 clones est présenté ci-dessous.
Q3
- Comment peut on interpréter la carte de restriction présentée ci-dessus?
Q4
- Que faut-il faire pour la suite des expériences ?
Etape 4: définition d'amorces
Pour valider les clones positifs, une vérification par séquençage de la zone d'intérêt est nécessaire. Il faut donc disposer d'amorces (oligonuclétoides) adéquats pour le séquençage. Différents logiciels sont disponibles pour définir des amorces dont PrimerBlastqui présente l'avantage de combiner un logiciel de définition de primer disponible sur le Web (Primer 3), à une analyse de type Blast de vos primers sur les banques du NCBI pour en vérifier leurs spécificités.
Utilisation de Primer-Blast Copier-coller la séquence du vecteur pVX_CBEL au format fasta, dans le grand cadre blanc en haut de page Définir dans "range" les positions de la zone à amplifier Définir la taille de l'insert à amplifier (sans importance pour le séquençage, indispensable pour une PCR ou qPCR) Définir le Tm de vos primers (paramètres par défaut) Sélectionnez les banques à utiliser pour l'analyse Blast (Organism : all, database : nr)
Modification du format de séquences (fasta, texte..) Utilisez le logiciel SeqRet Copier-coller la séquence pVX_CBEL générée avec pDRAW32 dans le cadre Choisir le format de sortie "pearson/fasta" Sauvegarder votre séquence pVX_CBEL, format fasta, pour l'utilisation dans Primer_Blast
Q5
- Définir un couple d'amorce permettant le séquençage de la zone d'intérêt en 5' et 3' (jonction vecteur/insert ; insert CBEL ; jonction insert / vecteur) de pVX_CBEL
Etape 5: séquençage
Quatre clones dont le profil de digestion ClaI/ NotI est en accord avec la carte de restriction pVX_CBEL, vont être séquencés. Les séquençages peuvent s'effectuer sur le site de la plateforme technique Génotoul de Toulouse Midi-Pyrénées ou via d'autres prestataires.
Les résultats de séquençage sont généralement téléchargeables sur votre ordinateur au laboratoire. Avant toute analyse de séquence il est impératif de vérifier la qualité du séquençage (Phred) et de corriger votre séquence par lecture des chromatogrammes. Vous trouverez différents sites internet vous permettant de réaliser ces opérations, comme celui de l'Université du Michigan
Etape 6: analyse des résultats
Avant de continuer les expériences à la paillasse, il est impératif de BIEN VERIFIER votre clonage et donc que l'insert cloné correspond bien à l'ADNc de CBEL, que l'insert est bien orienté et positionné dans le vecteur pVX entre les sites de restriction ClaI et NotI.
Analyse des resultats de sequencage par Blast Deux clones independants ont été sequencés (IPM_60 et IPM_61), les sequences sont disponibles sur ce lien au format fasta
Q6
- Interprétez la sortie Blast, sur la banque nr, de IPM_60 et IPM_61
- Avec quelle amorce la séquence IPM_61 a-t-elle été obtenue ?
- Que pouvez vous conclure ?
Compte-tenu des résultats précédents, il est nécessaire de réaliser un alignement multiple entre IPM_60 et IPM_61, en manipulant préalablement IPM_61.
- Inverser et complémenter IPM_61. Vous pouvez utiliser le logiciel online reverse complement
- Renommer votre nouvelle séquence IPM_61rev.
- Réaliser un alignement entre les 2 séquences, en utilisant le logiciel online multalin
- Verifiez la présence de l'insert CBEL (codon start :: codon stop), des sites de restriction (ClaI:5'-ATCGAT-3' et NotI:5'-GCGGCCGC-3'), du vecteur de clonage PVX.
Q7
- Que pouvez-vous conclure ?